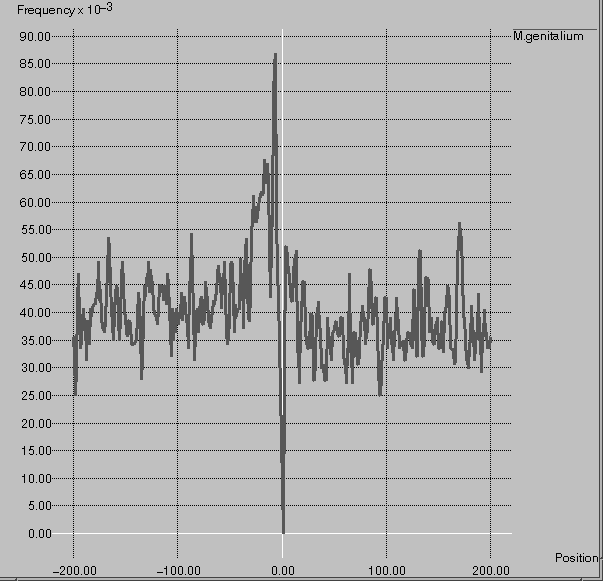
塩基配列パターン“ATG”は開始コドンと呼ばれる暗号として使われている。したがってこれと同じパターンが開始コドンの周辺に存在していると、蛋白質の合成が正しく行われなくなるため、進化的に開始コドン周辺のATGには淘汰圧がかかっているはずである。我々は様々な生物でATGの頻度が開始コドン周辺でどのように変化するか、解析した。その結果、真核生物と原核生物では頻度の変化が大きく異なることが判明した。これは原核生物ではリボソームと呼ばれる細胞小器官が直接mRNAに結合するのに対して、真核生物ではリボソームがmRNA上をスライドするという仮説をサポートする。
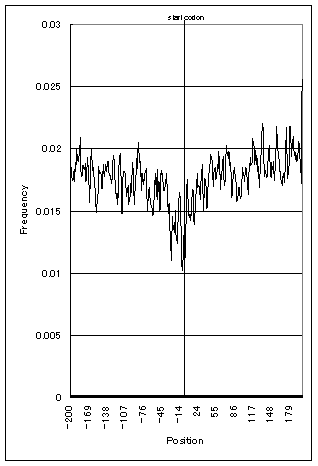
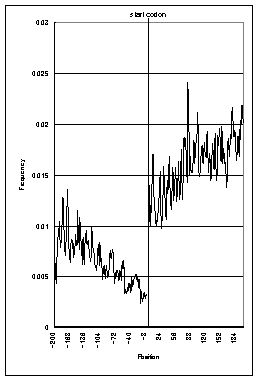 ・
・