森基金研究成果報告書
研究者育成費(修士課程)
E-CELLにおける細胞記述言語-EMLの開発
慶應義塾大学 大学院政策・メディア研究科 修士課程2年
櫻田剛史
概要
本研究では汎用細胞シミュレーションソフトウエアE-CELLにおける細胞モデル記述言語(E-CELL Model description Language: EML)の開発を行った.現在,E-CELLプロジェクトでは,より大規模で精密なモデル構築することを目指し,大幅に機能を拡張したE-CELL3の開発を行っている.E-CELL3においては,これまでに比べより大規模な細胞モデルのシミュレーションが行えるようになる.従来のE-CELL1におけるモデル構築環境においては,モデルが大規模で複雑さを極める今後のE-CELL3利用環境には対応できず,より効率的な細胞モデル構築手法の開発が必要不可欠であった.そこで,本研究ではこのうち特にモデルの記述方法に着目し,細胞モデリングに特化した記述言語EMLをXML技術をベースとして設計・実装した.
研究成果
本研究はE-CELLシミュレーション環境バージョン3の開発の一部として主に,以下に列挙する問題に取り組んだ.
EMLの仕様設計とXML Schemaによる文章型定義の記述
新しいモデル記述言語EMLの仕様設計を行った.EMLはE-CELL3のコア上で構成されるモデルオブジェクトを永続化した1対1対応のものとして存在している.
モデルを構成する上で基本となるエレメントはStepper, System, Variable, Process, Propertyの5つがある.これは基本的にE-CELL3コアシステムで形成されるシミュレーションモデルと対応する(図 1).
設計したEMLの仕様を元に,XMLの文章型定義(DTD)をXML Schemaを用いて記述した.
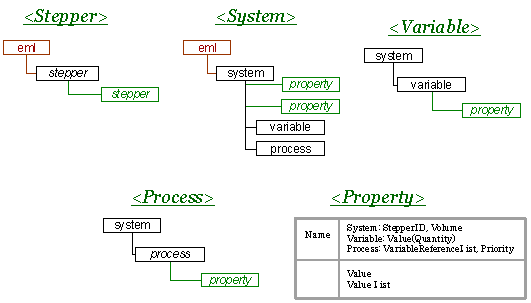
図 1 EMLを構成する要素
EMLに直接関連するソフトウェアの開発
emllib
EMLで記述されたモデルをロードして,コアシステムに渡せるようにするライブラリーであり,Python言語を用いてプログラミングされている(図 2).モデルのデータ構造としては,DOMを用いており,現時点では,E-CELL3上のモデルオブジェクトの永続化された形としてEMLが存在する.
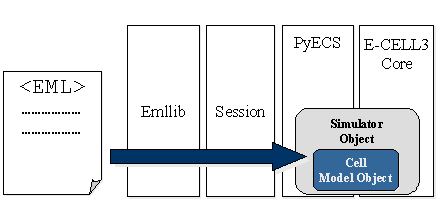
図 2 EMLのロード
em2eml/eml2em
XMLのサブセット言語であるEMLはヒューマンインターフェイスが貧弱なため,直接人間が記述・編集することは効率が悪く適当でない.そこで,EMLを生成するための1段メタなレベルでの編集を可能とする言語を設計として,em形式を設計し,emとEMLの間の相互の変換ツール(図 3)を開発した.

図 3 EM2EML/EML2EM
eri2eml
E-CELL3の環境では,モデルはすべてEMLを基本として記述,保存されることになったが,これまでのE-CELL1との完全な上位互換をとることはできず,E-CELL3においては,E-CELL1で構築したモデルをそのまま使うことはできない.
E-CELL1でモデル蓄積したユーザーができるだけ容易にE-CELL3の環境へ移行できるように,E-CELL1の.eri形式を自動でEMLに変換するコンバーターとしてeri2emlを作製した(図 4).
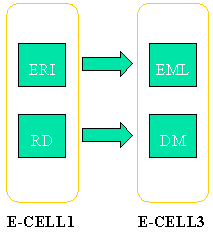
図 4 モデルデータのコンバート
GraphVizを用いたパスウェイの可視化
取り扱うモデルの規模が大きくなるにつれて,モデリングを行う人間の認識能力が追いつかなくなる.このようなときに,そのモデルがテキストのリストの形ではなく,図形を使って示された形でも見ることができれば,認識できるモデルの規模がより大きくなることが期待できる.
GraphVizは米AT&T社で開発された汎用のグラフ描画ソフトウェアである.今回はEMLをemllibを使ってパースしたあと作られるDOMツリーからGraphVizが理解するデータファイルに変換するプログラムを作成した.簡単なモデルでの描画例を図 5に示す.
自動描画は難しく,できるだけ見やすいマップが描かれるようにパラメーターをチューニングする必要があった.
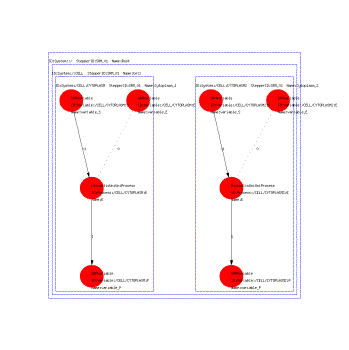
図 5 GraphVizを用いたパスウェイの可視化
課題
今後のE-CELLシステム,モデリング環境の開発に関して検討を要する事柄については次に上げる項目がある.
XMLレベルでのエラー検出の有効性とその限界について
まず,整形式(well-formed)としてのエラーチェックはモデルデータをロードするパーサーの時点でかける必要があり,この時点でのタイプミスなどXMLになっていないエラーを検出することには意味があることが実証済みである.次に,より厳密な妥当(valid)性の検証行った場合について考えると,現在のEML仕様によって拘束される文法でのエラーチェックはモデリング環境においては意味が薄く,モデル自体が持つ構造の意味(セマンティクス)としてのモデルのエラーチェックはXML Schemaだけでは限界があり,もう一段階モデルの中身を解析した上で評価を行うことが必要になるだろう.
データベースとの連携について
今日,細胞シミュレーション研究の対象の規模は大きくなる一方であり,そのモデリングには,データベースとの連携が必要不可欠になってくるはずである.しかし,今のところモデリング上のデータベースの利用は,個別のモデリングプロジェクトによって独自にすすめられおり,汎用的とは言えない面もある.今後,ふつうにモデリングにデータベースを使うようになるのは確実であるが,そのためにもE-CELL側に統一的にアクセス可能なインターフェイスを容易すべきではないだろうか.
SBMLとの相互互換性について
現在,SBML開発のための国際コンソーシアムには世界の有力なシミュレーターが数多く入っている.E-CELLもその一つであるが,SBMLをサポートして直接モデルをロードできるようにすることにはE-CELLの汎用化の目的では非常に有益である.現在,基本的な構造,モデルを記述するものとしてLevel1が勧告されている.また,今年度の活動により,それを拡張したLevel2を開発した.しかし,このLevel2の中では,E-CELLチームなどが提案したモジュラリティに関する拡張は取り込まれず,来年度のLevel3に向けて議論を続けることになった.
SBMLで記述したモデルのE-CELL SEへのインポートについては,コンソーシアム,コミュニティよりSBMLパーサライブラリーを提供してもらうことが可能になれば,比較的容易にE-CELL3への実装は可能だろう.
発表実績
学会ポスター発表
• ICSB2002 (International Conference Systems Biology 2002), (2002, Stockholm,Sweden)
• US-Japan JointWorkshop on Systems Biology of Useful Microorganisms (2002,Tsuruoka, Japan)
• ISMB2002 (Intelligent Systems for Molecular Biology 2002), (2002, Edmonton,Canada)
• 第24回日本分子生物学会年会(2001 年, 横浜)
• 21世紀の先端生命科学先端生命科学研究所オープン記念シンポジウム(2001 年,鶴岡)
論文
・The Systems Biology Markup Language (SBML): A Medium for Representation and Exchange of Biochemical Network Models
M. Hucka, A. Finney, H. M. Sauro, H. Bolouri, J. C. Doyle, H. Kitano, A. P. Arkin, B. J. Bornstein, D. Bray, A. Cornish-Bowden, A. A. Cuellar, S. Dronov, E. D. Gilles, M. Ginkel, V. Gor, I. I. Goryanin, W. J. Hedley, T. C. Hodgman, J.-H. Hofmeyr, P. J. Hunter, N. S. Juty, J. L. Kasberger, A. Kremling, U. Kummer, N. Le Novere, L. M. Loew, D. Lucio, P. Mendes, E. D. Mjolsness, Y. Nakayama, M. R. Nelson, P. F. Nielsen, T. Sakurada, J. C. Schaff, B. E. Shapiro, T. S. Shimizu, H. D. Spence, J. Stelling, K. Takahashi, M. Tomita, J. Wagner, J. Wang. In press, Bioinformatics, 2003.
・櫻田剛史,E-CELL3における細胞シミュレーションモデル記述言語の設計と実装,修士論文,2002,慶應義塾大学
謝辞
本研究はE-CELL シミュレーション環境バージョン3開発の一環として行われた.特に開発チームリーダーの高橋恒一氏には,研究のあらゆる場面での助言を頂いた.また,鈴木良介氏,山田洋平氏,北山朝也氏,井元淳氏らシステム開発メンバーの皆様からも様々な場面でご助力・助言を頂いた.
また,冨田研究室のスタッフの皆様には研究の様々な場面で助言,助力を頂いた.とりわけ,内藤泰宏専任講師,中山洋一専任講師には適切な指導をして頂いた.
そのほか,数が多くこの場にすべて名前を挙げることができないが,E-CELL プロジェクトメンバーすべてを含む,お世話になった方々へこの場を借りて厚くお礼を申し上げる.
最後に,私に研究の機会を与え,常に合理的でかつ適切な助言をして頂いた冨田勝教授へ深く感謝を申し上げて本論文の結びとしたい.
本研究は,2002年度森泰吉郎記念研究振興基金研究者育成費(修士課程)の支援を受けて行われた.