2003年度森基金
研究成果報告書
課題名:E-Cell モデリング環境の開発
2004年2月27提出
政策・メディア研究科
後期博士課程1年
櫻田 剛史
緒言
研究の概要
E-Cell Systemは特に細胞シミュレーションに特化した機能を実現したモデリング、シミュレーションのためのソフトウェアプラットフォームである。構成するモジュールとして、
u
E-Cell Simulation Environment
version 3 数値シミュレーションのためのソルバー、およびデータ構造を持つ基盤ソフトウェア
u
E-Cell Modeling Environment 細胞モデル構築のためのエディター、データベースなど
u
Analysis toolkit 未知パラメーターの推定、分岐解析を行うソフトウェア
の3つで構成される
E-Cell Systemにおけるモデルデータは、主にEML (E-Cell
Model description Language)によって記述される。これは、XML形式のデータであり、これ自体はユーザーが編集するには適さない。昨年度までに、テキスト形式からEMLを生成するツールを開発したが、より効率の良いモデリングのため、グラフィカルユーザインターフェイス(GUI)ベースでのモデルエディターを開発した。今年度はその設計と基本的なフレームワークの実装を行った。
大規模、複雑な細胞モデルを編集するために,モデルエディターの設計では、2つの編集用ビューを用意した。具体的にはGUIベースで細胞モデル中の反応パスウェイを表示・編集する(パスウェイビュー)、E-Cell3の中で表現されるオブジェクトとしてのモデルの編集する(オブジェクトツリービュー)を持つ。
現在までに、パスウェイビューを除いた、ユーザーが試用できるベータバージョンをパッケージとしてリリースしている。
本研究の目的
本研究では、E-Cell Systemにおけるモデリング環境の開発を主目的とし、特に、モデルエディターの設計と開発に取り組んだ。
研究成果
要求分析と設計
要求分析
ソフトウェアの内部設計と実装に先立って、モデリング環境に対する要求分析を行った。前バージョンであるE-Cell 1を対象とした、これまでのモデリング環境の問題点を調査したところ、以下の項目について問題点が挙げられた。
1. 検出しうる明らかなエラーの検出などモデルデバッグ機能の不足
Ø タイプミスの検出
Ø IDの不一致の検出
Ø 単位の不一致、変換ミスの検出
2. GUIベースでのモデリング機能の不足
Ø 代謝、シグナル伝達などのネットワークの描画によるものが求められる
3. C++による計算モジュールの作成
Ø ユーザーにC++の知識を要求することによってモデリングの難易度を必要以上に上げているケースが存在
このうち1.については、前年度までのEMLおよびEMモデリングツール(図1)によってある程度の改善が行われ、引き続きこの部分については、EMLモジュールの担当として開発が継続されている。3.については、共同研究者の石田氏(政策・メディア研究科)によって、数式、あるいはスクリプトを直接EML中に記述してC++での計算モジュールを不要とするPythonProcess, ExpressionFluxProcessが開発されている。
今回は2.についてこの問題を改善すべく、モデルエディターの仕様設計を行った。
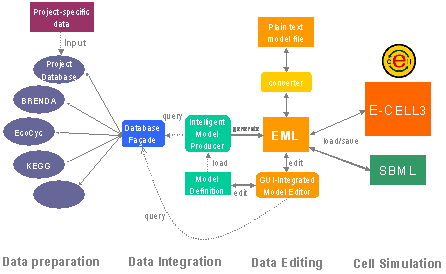
図 1 E-Cellモデリング環境
モデルエディター仕様設計
解決すべき問題については前節に挙げた通りであるが、これを解決するために具体的なモデルエディターの仕様設計を行った。その仕様の特徴としては次のようなものが挙げられる。
A) E-Cell SEでのオブジェクトベースでのシミュレーションに対応可能なオブジェクトベースのモデリングを指向
B) EML (E-Cell Model description Language)をロード/セーブ可能
C) パスウェイビュー、パスウェイエディターの機能を装備
A) E-Cell SEでのオブジェクトベースでのシミュレーションに投影可能なオブジェクトベースのモデリング
E-Cell SE (Simulation Environment)は特に細胞シミュレーションに特化したシミュレーション基盤としての開発中のものであるが、この特徴の一つとして、オブジェクトベースでのシミュレーションが挙げられる[1]。これは、これまでの生化学シミュレーションで用いられた式ベースでのシミュレーション、つまり連立微分方程式を数値的に解くことによるシミュレーションとは異なり、シミュレーションモデルをオブジェクトとして取り扱いそれぞれが持つプロパティ、イベントとしてシミュレーションを行うものである。
E-Cellモデルエディターにおいても、このオブジェクトベースのシミュレーションにそのまま対応した、編集モードを用意した。これは、その外観としては、E-Cell SEでのGUIセッションモニターにおけるEntityListWindow, StepperWindowとほぼ同じモデルオブジェクトの表示方法を持ち、編集を可能にするものである(図2)。
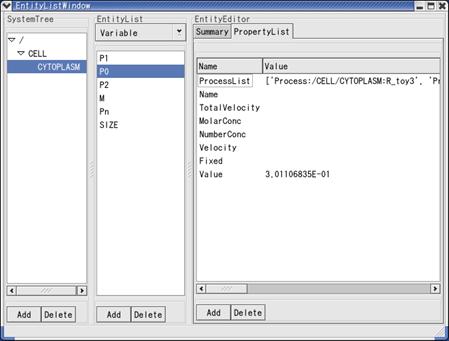
図 2 EntityListWindow
B) EML (E-Cell Model description Language)をロード/セーブ
前節Aで解説したように、E-CellモデルエディターはE-Cell SEと同等のモデル構造を編集可能であるが、これを具体的に保証するものはE-Cell SEにおけるモデリング言語であるEMLである。モデルエディターはE-Cell SEにおけるシミュレーションオブジェクトを永続化した存在であるEMLを介してデータの交換を行う。
これに関連してモデルエディターにおけるモデルデータ構造として、ModelStoreクラスを設計した(図3,4)。
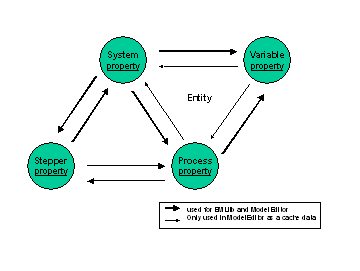
図 3 ModelStoreそれぞれのオブジェクトの関係

図 4 ModelStoreクラスAPIリスト
C) パスウェイビュー、パスウェイエディターの機能
代謝、シグナル伝達のモデルを人間が理解する上で有益な表現法としてパスウェイ、ネットワークをグラフィックで表示、編集する機能が挙げられる。E-Cell モデルエディターにおいて、パスウェイビューは前節Aで解説したE-Cell SEでのオブジェクトの存在をそのまま表現することを目的として設計された(図5)。
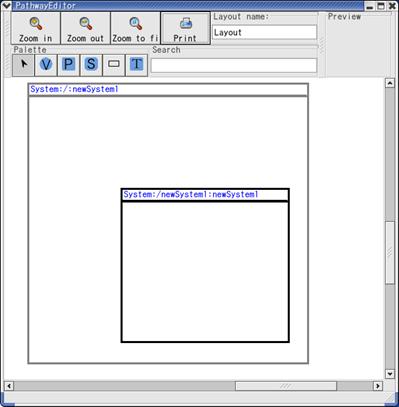
図 5 パスウェイエディター
モデルエディターの開発
開発環境として、RedHat Linux 9 および、Fedora Core 1を用い、GUIツールキットには、GTKとGnomeディスクトップ環境を用いてPythonスクリプト言語を用いてコーディングを行った(図6)。
現在までに、e-cell-3.1.100としてモデルエディターを同梱したパッケージをWebサイト[2]でリリースしている。
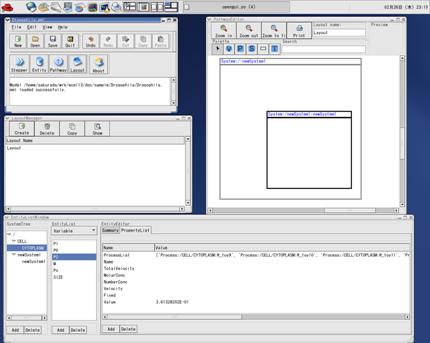
図 6 モデルエディターのスクリーンショット
残された解決すべき課題
今年度に本格的な開発に取り組んだモデルエディターについて、これまでに実際にユーザーが使いうるベータバージョンをリリースすることができた。ただ、現時点では、パスウェイエディターの実装が未完成であり、またユーザーに対するドキュメンテーションが不十分である。これらついては継続課題とする必要がある。
謝辞
本研究課題について、特にお世話になった皆様に厚く御礼申し上げる。
スーパーバイザー:冨田勝教授
研究アドバイザー:中山洋一専任講師、高橋恒一氏(慶應義塾大学SFC研究所)
共同研究者:Gabor Bereczki氏(慶應義塾大学先端生命科学研究所)、石田達也氏(政策・メディア研究科)
本研究は2003年度慶應義塾大学森泰吉郎研究者育成費(博士課程)、および、平成15年度慶應義塾大学21世紀COEプログラム"システム生物学による生命機能の理解と制御"の助成を受けて行われた。
学会発表等一覧
ポスター発表
Interoperability
of E-CELL3 and SBML
Takeshi Sakurada, Nayuta Iwata,
Kouichi Takahashi, Masaru Tomita, The First IECA Conference on Systems Biology
of E.coli (IECA 2003),
E-Cell system
version3 大規模細胞シミュレーションのための統合環境開発
高橋恒一, 櫻田剛史, 海津一成, 北山智也, Satya
Arjunan,
石田達也, 伊東大輝, 杉本昌弘, 小森隆, 太田聖治, 冨田勝
ORF2003,
Development of
E-Cell Modeling Environment Takeshi
Sakurada, Kouichi Takahashi, Masaru Tomita, International Symposium of
21st-Century COE Program, Keio University Understanding and Control of Life's
Function via Systems Biology, Yokohama, November 2003
E-Cell3:大規模細胞シミュレーションのための統合ソフトウェアプラットフォーム
高橋恒一,櫻田剛史,海津一成,北山朝也,Arjunan Satya,石田達也,伊東大輝,杉本昌宏,小森隆,太田聖治,冨田勝, 第26回日本分子生物学会年会, 神戸, 2003年12月
ソフトウェアデモンストレーション
E-Cell System
Version 3: A Software Platform for Integrative Computational Biology Kouichi Takahashi, Takeshi Sakurada,
Kazunari Kaizu, Tomoya Kitayama, Satya Arjunan,
Tatsuya Ishida, Gabor Bereczki, Daiki Ito, Masahiro Sugimoto, Takashi Komori, Ohta Seiji, Masaru Tomita, GIW2003, Software Demonstration,
Yokohama, December 2003
参考文献
[1] "Computational challenges in cell simulation", K. Takahashi, K. Yugi, K. Hashimoto, Y. Yamada, C.F. Pickett, and M. Tomita, IEEE Intelligent Systems, 17(5):64-71, (2002).
[2] E-Cell Project http://www.e-cell.org