森基金成果報告書(研究育成費)
E-CELLにおけるSBML(Systems Biology
Mark-up Language)のサポートモジュールSBML Importer開発
慶応義塾大学 政策・メディア研究科
修士1年 石田達也
1.はじめに
現在、大規模な細胞や代謝回路などのモデル化がシステムバイオロジーの分野で非常に重要視されている。これらのモデルをコンピュータ上で表現するため、多くの研究者がシュミレーションソフトウェアなどを開発している。E-Cellグループもその内の一つである。しかし、細胞をモデリングする方法は個々のソフトウェアで独立しており、ソフトウェア間での互換性がなかった。また、生命システムの解析手法も多岐にわたっており、一つのソフトウェアで全てをカバーすることは現実的ではない。
そこで提案されたのがSBML( Systems Biology Mark-up Language)である。SBMLはXMLベースの生物モデル表現体系であり、多くの細胞シミュレータのデファクトとなっている。SBMLは2001年3月にLevel1がリリースされ、2003年7月にLevel2がリリースされました。現在も開発段階にあり、Level3に向けて多くの議論が交わされています。このSBMLをサポートしているソフトウェアは今現在40近くあり、SBMLの標準化がどれだけ必要であるかがわかるだろう。SBMLをサポートすることにより、他のソフトウェアとのモデルの交換、比較などが可能となり、モデリングにおいて視野も広がると考えられる。
今期、E-Cell3がSBMLをサポートするためのモジュールを作成した。SBMLサポートには、SBMLのモデルをインポートできる機能とE-Cellのモデルをエクスポートできる機能が必要であるが、E-CellのモデルをエクスポートするにはいくつかのE-Cell独自の解析手法をSBMLで表現することが難しいため、先にインポートするためのモジュールを作成した。
2.SBML( Systems Biology Mark-up Language)
SBML Level2は図1のような継承階層構造をとっている。矢印の元が継承されている子であり、指されているのが親である。SBMLをサポートするにはこれらの要素をすべてE-Cell3の要素に変換する必要がある。

図1:SBMLの主なデータの継承階層構造のUMLダイアグラム
コマンド
SBML Importerは次のコマンドで使うことができる。
% ecell3-sbml2eml sample.xml
コマンドはecell3-sbml2emlであり、その後にSBMLで書かれたモデルファイルを指定する。このコマンドにより、カレントディレクトリにSBMLモデルファイルと同名のEMLモデルファイルが生成される。
3.研究成果
今現在、E-Cell3がSBMLをサポートしている機能を図3に示す。
|
SBML Component |
サポート状況 |
|
Compartment |
○ |
|
Parameter |
○ |
|
Species |
○ |
|
Rule |
× |
|
Reaction |
○ |
|
Event |
× |
|
UnitDefinition |
○ |
|
FunctionDefinition |
× |
図3:サポート状況
今回、RuleとEventはサポートすることができなかった。RuleはlibSBMLが機能を増やすまで、サポートすることができない。これはlibSBMLの開発者と協力し、機能拡張を行っていくつもりだ。Eventはdelayという要素をサポートしていない。FunctionDefinitionはSBML Importerの中でマクロ展開すれば良い。
・PythonProcessの改良と新たなProcessの作成
E-Cellはシミュレーションをする際に、ユーザがアルゴリズムをプラグインとして作成し、それを使ってシミュレーションする方法をとっている。PythonProcessは、Kineticの情報をモデルファイルに書き込むことにより、新たなプラグインプログラムを作成する必要がなくなる。しかし、PythonProcessはシミュレーションの際に実行速度が非常に遅く、プラグインプログラムでシミュレーションした場合の10倍から50倍の速度低下があるという欠点が見られた。そこで、コードを改良し速度向上を試みたがPythonのParserが遅いことがわかり、PythonProcessに代わる新たなプラグインプログラムを作成することにした。そこで作成したものが、ExpressionProcessである。ExpressionProcessでは、そのプラグインプログラムの中でC++がコンパイルを行い、新たな仮想的コードを作成し計算するプログラムである。これを使いシミュレーションしたところ、プラグインプログラムを作成する場合より約2倍程度遅いものであったため十分使い物になると考え、これをSBMLをサポートするプラグインプログラムとして使うことにした。下の3つのProcessはExpressionProcessBaseを継承しているProcessである。
ExpressionAlgebraicProcess
代数式を計算するためのProcessである。0 = g(t,x)で表された式をExpressionに代入し、その計算結果をsetActivityする。これを使用するためにはDAEStepperを使う必要がある。DAEStepperは連続的なProcessと離散的なProcessの混在を可能にするStepperである。現在DAEStepperはAlgebraicProcessをサポートしていないので、FixedDAE1Stepperを使う。
例: A * B = 1
Stepper FixedDAE1Stepper( DAE1) {}
Process ExpressionAlgebraicProcess( Process1 )
{
Stepper DAE1;
Expression
“A * B - 1”;
VariableReference [S0 :.:A 1] [S1
:.:B 1];
}
ExpressionAssignmentProces
化学量論係数が0以外の値にsetValueするProcessである。化学量論係数が0以外の物質にExpressionで計算された値とその化学量論係数を掛けた値を代入する。
Process ExpressionAssignmentProcess( Process1 )
{
Expression
“S0.MolarConc * Parameter0 + Parameter1”;
VariableReference
[S0 :.:Value1 1]
[Parameter0 :.:Value2 0] [Parameter1 :.:Value3
0];
}
ExpressionFluxProcess
計算結果をsetFluxしてくれるProcessである。KineticLawの場合、E-Cell3は分子量を扱うため、計算した値に自分自身のシステムのSIZEとアボガドロ数を掛け合わせてsetFluxする必要がある。
Process ExpressionFluxProcess( Process1 )
{
a 2;
Expression
“a * S0.MolarConc * P0.MolarConc + P1.MolarConc *
self.getSuperSystem().SizeN_A”;
VariableReference
[S0 :.:Value1 -1] [P0 :.:Value2 1] [P1 :.:Value3 1]
・モデル比較
次にSBML Importerのテストを行った。SBMLで書かれたモデルを他のシミュレーションソフトウェアとE-Cell3とで比較した。使用したソフトウェアはGepasiである。GepasiはPedro Mendesによって書かれたフリーソフトウェアであり、以下[ http://www.gepasi.org/ ]よりダウンロードすることができる。
モデルはSBMLのモデルレポジトリのものを使い、Gepasiとの比較を行った。モデルはhttp://www.sbml.org/models/からダウンロードすることができる。下のモデルは例として挙げた概日振動の最小モデルであり、SBMLのモデルレポジトリから得られたものである。図4はE-Cell3へインポートされたモデルであり、図5はGepasiへインポートされたモデルである。
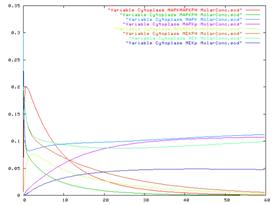
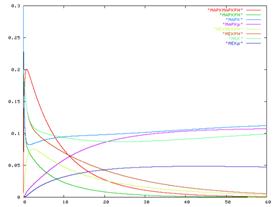
図4:E-Cell3にインポートされたモデル 図5:Gepasiにインポートされたモデル
図4と図5を比較してもわかる通り、同じ挙動を示している。この例や他のSBMLサンプルモデルをE-Cell3とGepasiで比較したところ、どれもが挙動が一致した。これによりSBML
Importerの確証性が証明できた。
4.課題
今回はSBML Importerを作成した。これにより、他のシミュレーションソフトウェアとの互換性を持ったと言えるだろう。しかし、E-Cell3からSBMLに変換するSBML
Exporterはまだ開発されていない。今後、E-Cellグループで開発されているモデルを世界の研究者に示すためにも、SBML
Exporterを使える環境が必要となるであろう。そのためにSBML Exporterを開発し、モデルを自由に交換できる環境を作っていこうと思う。また、新たなモデルをSBMLのモデルレポジトリにエクスポートし、E-CellグループがSBMLとの互換を持ち、モデルの交換が自由な環境が整ったことを示していきたい。
5.謝辞
本研究を進めるにあたり、E-Cell3開発グループの方々に多くの助言をかり、特に海津一成氏、櫻田剛史氏、高橋恒一氏には多くの助言、サポートをして頂きました。この場を借りて厚くお礼を申し上げます。また、このような研究環境を与えて頂いた冨田勝教授に感謝の意を深く表します。
8.参考文献
[1] “libsbml Developer’s Manual”
[2] “libsbml API Reference Manual”
[3] “Systems Biology Markup Language (SBML) Level 1:
Structures and Facilities for Basic Model Definitions” Michael Hucka,
Andrew Finney, Herbert Sauro, Hamid
Bolouri, Systems Biology Workbench Development Group
JST ERATO Kitano Symbiotic Systems Project
[4] ”Systems Biology Markup Language (SBML) Level 2:
Structures and Facilities for Model Definitions” Andrew Finney, Michael Hucka,
Systems
Biology Workbench Development Group ERATO Kitano Symbiotic Systems Project