
2003年度 森基金報告書
静的/動的ハイブリッドアルゴリズムを用いた大腸菌主要代謝経路のモデリング
政策・メディア研究科2年 冨田勝研究会所属
石井伸佳 (学籍番号 80231116) nishii@sfc.keio.ac.jp
1.はじめに
本研究では、微生物のComputer aided designに利用できるツールとして、大腸菌の全代謝モデルの構築を目指している。このための最初のステップとして、主要な代謝産物流束が存在すると考えられる、essential gene(生存に必須な遺伝子)を含む代謝系セットのモデリングを行った。
2.成果
現在までに知られている大腸菌のessential geneのうち、代謝に関連するものを調べ、これらを含む代謝系セットをExPASy, EcoCyc, KEGGを参照して選別した。選別した代謝系セットにはTable 1に示すものが含まれ、190反応、153 酵素、212 化合物からなる。
Table 1 選抜されたモデル化対象代謝系
| glycolysis, fermentation, TCA cycle, pentose-phosphate pathway, purin and pyrimidine nucleotides biosynthesis, fatty acid and phosphatides biosynthesis, cell wall components (diamino pimerate and amino sugars) biosynthesis, isoprenoid and ubiquinone biosynthesis, heme biosynthesis, respiratory chain |
代謝系セットのリストと、遺伝子データベースを利用して大規模な代謝の化学量論モデルを作成するGEM
System[1]の出力ファイルを照合し、基本モデルを作成した。
基本モデルをダイナミックシミュレーションが行えるように変更するには、各酵素のキネティックパラメータが必要である。しかし、全ての酵素についてキネティックパラメータを取得するのは現実的ではないので、動的/静的ハイブリッドシミュレーションアルゴリズム[2]を適用して動的に表現すべき酵素の数を削減することにした。
この場合、モデル中の律速酵素を同定する必要がある。そこで、文献データベース(PubMed)を検索し、アブストラクト文に含まれる律速酵素を抽出した。873(酵素番号が判明したのは589)の律速酵素が得られた。
律速酵素リストに含まれる全ての酵素が大腸菌において実際に律速となっているとは考えられないため、律速酵素の絞り込みを行った。PubMedのアブストラクト文での出現頻度およびEcoCyc,
BRENDAに記載されているエフェクターの数に基づき律速酵素の重要度をランキングし、出現頻度10回以上またはエフェクターの数が3つ以上のものに限って基本モデル内の所在を確認したところ、26酵素まで削減することが出来た(Table
2)。
Table 2 モデル対象代謝系セットに含まれる重要な律速酵素

更に、モデル内で端点となる酵素、慶應義塾大学 先端生命科学研究所 吉野昌孝教授によって詳細なデータが測定された解糖系酵素[3]について動的に表現することとし、計93反応を動的部位として、静的/動的ハイブリッドアルゴリズム[2]を適用した。動的部位の酵素反応速度論的情報はBRENDAより収集した。
作成したモデルにより、有酸素状態から無酸素状態へ条件が変化したときの代謝系の状態遷移をシミュレーションしたところ、モデルに含まれる約半数の酵素についてしか反応速度式を用いていないにも関わらず、TCA回路の活動が減少してATP生産能力が低下し、乳酸やエタノールの生産速度が上昇する現象を表現できた(Fig.
1)。
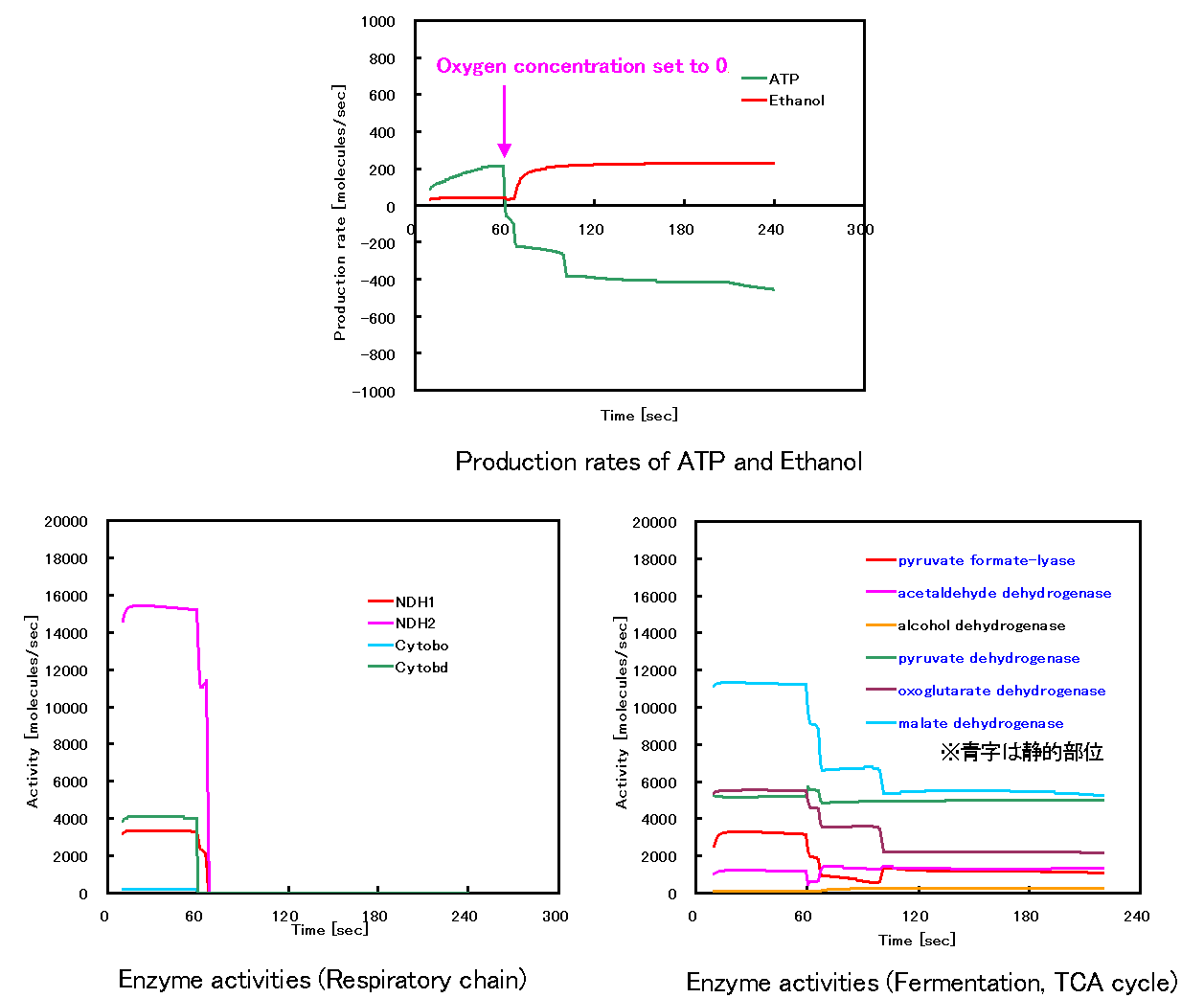
Fig. 1 静的/動的ハイブリッドアルゴリズムを使用した大腸菌モデルによるシミュレーション
なお、以上のモデルでは文献調査した律速酵素を動的部位として用いたが、今後、精度の高いモデルを構築するには、対象となる生物種・生理条件における律速酵素を実験的に同定するのが重要である。そこで、このための基礎として酵素活性の時系列データが得られたとき、遺伝的アルゴリズム(GA)によって律速酵素を同定する技術を開発した(Fig 2, Table 3)。
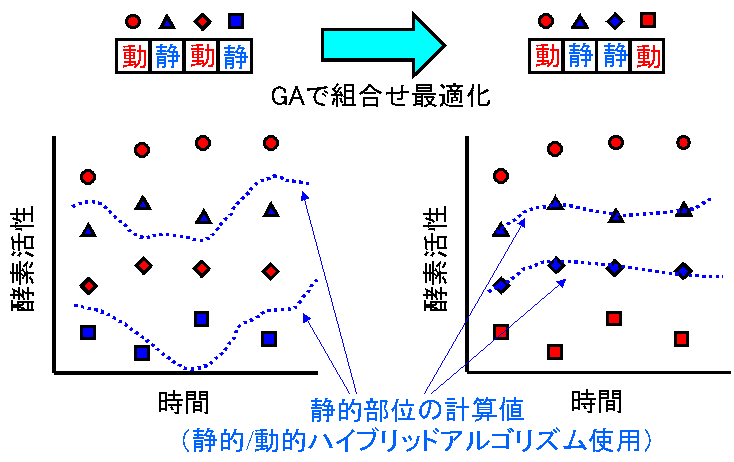
Fig. 2 GAによる律速酵素同定法 概念図
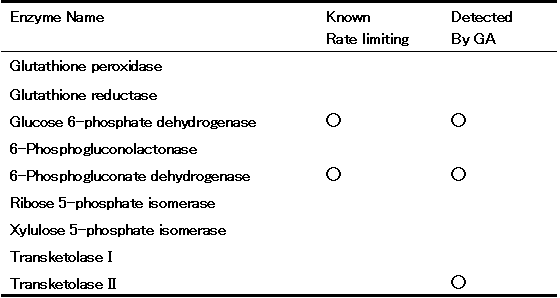
Table 3 ヒト赤血球モデル[4]の酵素活性時系列より検出した律速酵素
3.今後の予定
今後は、先端生命科学研究所にて各種の実験を行い、モデルの完成度を向上させることが課題である。代謝産物濃度の時系列実測データより酵素活性の時系列を求めて律速酵素を同定する技術を確立し、さらに実際に大腸菌において律速酵素を同定する予定である。また、それら律速酵素の詳細な反応速度パラメータ測定や、シミュレーションの実行や計算結果の検証に必要な代謝産物濃度の測定も行いたい。
4.参考文献
[1] Arakawa, K., Yamada, Y., Shinoda, K. (2002); Genome-based Large Scale
Modeling: GENESYS on G-language Genome Analysis Environment. 生命と情報, 9,
45-63 (2002)
[2] 中山洋一(2002); 細胞シミュレーションモデルを構築する.
蛋白質 核酸 酵素, 47,1956-1961
[3] Yoshino, M., Ogawa, T., Mori, H. and Tomita, M. (2003) "Kinetic analysis and regulatory properties of
glycolytic enzymes purified from archived clones of E. coli." Internal Workshop for
Escherichia coli., Awajishima
[4]中山洋一, 冨田勝(2000) "E-CELL システムを用いたヒト赤血球細胞シミュレーションと病理解析への応用" 遺伝子医学, 4, 49-53