In vitro virus(IVV)法を用いた選択的スプライシングの機能解析
慶應義塾大学大学院 政策・メディア研究科 柳澤 直宏
要旨
ヒトゲノムや完全長cDNAなどから得られたヒトの遺伝子数が当初の予想よりも大幅に少なかったことから選択的スプライシングが高等真核生物の多様性を生む重要な役割を担っていると考えられている.我々はまずタンパク質間相互作用部位を網羅的に検出できるin vitro virus (IVV)法のデータを網羅的に解析することで選択的スプライシングによって機能が変化する遺伝子を探索し,14の候補遺伝子を抽出した.更に候補遺伝子よりcassette型のスプライスパターンをもつFOS(ヒトの代表的な転写因子)に注目し,プルダウン法やIVV法を用いてcassette型の選択的スプライシングがタンパク質間相互作用(PPI)およびタンパク質-DNA相互作用(PDI)に及ぼす相互作用能の変化を確認した.また,RT-PCR法やcDNAライブラリを用いてFOSスプライスバリアントの転写産物の有無を確認した.この結果,選択的スプライシングがPDIおよび細胞内の局在に重要な役割を果たしていることが示唆された.
1 序論
1.1 研究の背景
近年の遺伝子研究においてヒトゲノムや完全長cDNAなどの配列情報が決定されるに従って遺伝子の数とその種類ついては生命科学における論点となっている.特に高等真核生物であるヒトの遺伝子数は生物種間の比較からヒトゲノム解読以前には10万個必要といわれていたが,解読後の現在では約2万5千個といわれている.この様にヒトの遺伝子数が当初の予想よりも少なかったことから選択的スプライシングなどの遺伝子発現機構の多様性が生体内で重要な役割を果たしていると考えられている.
1.2 選択的スプライシング
選択的スプライシングとは真核生物の細胞内において通常行われているゲノム配列から転写されたmessenger RNA(mRNA)配列のイントロンを切断・除去するスプライシングという過程の中で,スプライスパターンを変化させることで多様なmRNAを生み出す仕組みである.この仕組みによって多様なタンパク質が生み出されている.
また近年の研究で,ヒトの全遺伝子の40%から60%で選択的スプライシングが起こっていると推計されている(Mironov, A. A. et al. 1999; Kan, Z. et al. 2001; Modrek, B. et al. 2001; Modrek, B. et al. 2002).
今回の我々の研究は,選択的スプライシングによって相互作用が変化する領域を網羅的に抽出していることから,選択的スプライシングがタンパク質の機能の変化に影響を与えている遺伝子を効率よく抽出できる可能性が高い.またその機能変化を実験的に再確認することも容易であることから,これまでのexpressed sequence tag(EST)を用いた解析では見出せなかった新たな遺伝子や選択的スプライシングの機能を見出せる可能性をもつ.
1.3 In
vitro
virus(IVV)法
In vitro virus (IVV)法は慶應義塾大学理工学部柳川研究室の宮本悦子講師を中心に開発されたmRNAとそのmRNAから翻訳されたタンパク質を対応付けする新規技術であり(Miyamoto-Sato, et al. 2005),この手法を応用することによって,タンパク質間相互作用(PPI)を検出することが可能である.特に IVV法は,細胞を用いないため,細胞毒性の問題はなく,より網羅的なPPI解析に適している.
1.4 IVV法の大規模化
2006年度IVV法のロボット化(QIAGEN BioRobotを使用)が実現した.これにより同時に最大96ベイトに対するPPIをおよそ1ヶ月で検出することが可能となり,膨大なPPI情報が得られる様になった.従って,IVV法を用いた大規模PPI研究において大量のデータの解析を得意とするバイオインフォマティクスの重要性は高いといえる.
2 IVV法を用いた機能的な選択的エキソンを持つ遺伝子 の網羅的抽出
2.1 目的
IVV法では特定のベイトタンパク質と相互作用する領域を持つプレイタンパク質が抽出される.更に,プレイタンパク質に対応するmRNAの配列を決定することで,タンパク質間相互作用領域を特定することができる.
以上を踏まえ,IVV法によって検出されたタンパク質間相互作用部位に選択的スプライシングが関与する例を網羅的に抽出することを目的とした.
2.2 対象と手法
IVV法の大規模化(2006年度)および,それに先駆けて行ったIVVのマニュアル実験(2004年度)の結果を利用した.実験で用いた50種類のベイトタンパク質は転写因子および転写因子関連タンパク質として知られているものである.
まず,basic local alignment
search tool(BLAST)(Altschul, et al. 1997)を用いてASD(Thanaraj, et al. 2004)のHuman Release 1およびRelease 2とIVV実験によって得られた相列情報との相同性検索を行った.次にopen reading frame(ORF)領域を確認することで,選択的スプライシングによってタンパク質としての機能が変化する可能性を検証した.更に,以上のような流れで抽出されたプレイ遺伝子に対して組織特異的な発現の有無を確認するために,EASED(Pospisil, et al. 2004),ASAP(Lee, et al. 2003)などの選択的スプライシングデータベースも参照し候補遺伝子の抽出を行った.
表 1:使用した選択的スプライシングデータベース
|
Database
Name |
URL |
|
The Alternative
Splicing Database(ASD) |
http://www.ebi.ac.uk/asd/ |
|
Extended
Alternatively Spliced EST Database (EASED) |
http://eased.bioinf.mdc-berlin.de/ |
|
The Alternative
Splicing Annotation Project (ASAP) |
http://www.bioinformatics.ucla.edu/HASDB/ |
表 2:解析に使用したIVVデータ
|
使用したデータ |
個数 |
|
使用したベイト総数 |
67 |
|
使用したベイトタンパク質数 |
50 |
|
実験によって検出されたPPIのペア数 |
945 |
|
解析の対象としたプレイ配列数 |
2033 |
一つのベイトタンパク質に対して,タンパク質全長,タンパク質モチーフ,スプライスバリアントなどが作成されているため,使用したベイト総数と使用したベイトタンパク質数は異なっている.
2.3 結果
IVV法によって得られた2,033配列とASD Human Release 2に登録されている16,293遺伝子を用いて相同性検索を行い,更に,IVV法によって得られた配列と相同性が高い領域をもつ個々の遺伝子についてスプライスパターンを比較した結果,選択的スプライシングによってタンパク質間相互作用領域を失うあるいは相互作用領域をもつことで機能が変化すると考えられる候補が14遺伝子抽出された(図 1,表3).
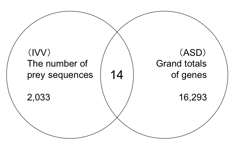
図 1:選択的スプライシングによって相互作用能が変化する遺伝子
表 3:抽出された14遺伝子
|
Bait |
Prey |
Description(Prey) |
|
MAX |
AIP |
aryl hydrocarbon receptor interacting protein |
|
FOS |
ATF2 |
activating transcription factor 2 |
|
JUN |
ATF2 |
activating transcription factor 2 |
|
NR2F6 |
CAMK2A |
calcium/calmodulin-dependent protein kinase (CaM kinase) II alpha |
|
NFKB1 |
ERCC6 |
excision repair cross-complementing rodent repair deficiency,
complementation group 6 |
|
NR4A1 |
FABP3 |
fatty acid binding protein 3, muscle and heart (mammary-derived growth
inhibitor) |
|
JUN |
FOS |
v-fos FBJ murine osteosarcoma viral oncogene homolog |
|
MAX |
HDAC5 |
histone deacetylase 5 |
|
FOS |
IDS |
iduronate 2-sulfatase (Hunter syndrome) |
|
MYC |
NAP1L1 |
nucleosome assembly protein 1-like 1 |
|
OPTN |
NDUFA12L |
NDUFA12-like |
|
ATF7 |
OCIAD1 |
OCIA domain containing 1 |
|
ETS1 |
PFDN5 |
prefoldin subunit 5 |
|
MAX |
RPL34 |
ribosomal protein L34 |
|
ETS1 |
SCOC |
Short coiled-coil protein |
|
SCHIP1 |
SCOC |
Short coiled-coil protein |
2.4 考察
2.4.1 FOSスプライスバリアント
本解析の結果,抽出された遺伝子は IVV法から得られたタンパク質間相互作用領域と選択的スプライシングの特徴から機能の変化が予想される.
特にASAPに登録されているFOSスプライスバリアントの選択的エキソンの位置とタンパク質間相互作用領域を比較すると,第3エキソンを欠くFOSスプライスバリアント(以後FOSΔbZIP1)はbZIPモチーフのうちDNAとの結合領域全域(basic region)およびロイシンジッパー(Leucine
zipper)の一部分を欠く(図 2).この変化に伴うc-fosタンパク質の相互作用能の変化を証明する為にはIVV法だけでなくc-fosと相互作用することが知られているc-junなどのタンパク質との一対一の相互作用を確認する必要がある.
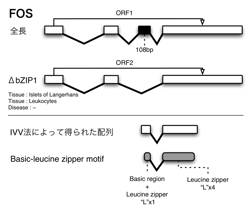
図 2:FOSスプライスバリアントの遺伝子構造
図の黒色部分は選択的エキソンを示しており,bZIP(灰色部分)の一部が変化する.
3 FOSスプライスバリアントの相互作用検証
3.1 目的
選択的スプライシングの役割を検討するため,抽出された14個の選択的スプライシングによって機能が変化すると考えられる遺伝子からFOSに注目しプルダウン法を用いて相互作用能の検証をした.
3.2 対象と手法
3.2.1 プルダウン法を用いたタンパク質間相互作用の検証
FOSΔbZIP1から転写・翻訳されたタンパク質はタンパク質間相互作用領域の一部を欠く.この変化により相互作用が知られているc-junタンパク質との相互作用を検証する.
3.2.3 プルダウン法を用いたタンパク質-DNA相互作用の検証
FOSΔbZIP1はタンパク質間相互作用領域の一部を欠く他, DNAとの結合領域全体を欠く.従ってPDIについてはON/OFFの変化が予想される.そこでPPIの変化に加えPDIの変化を実験的に検証した.DNAとの相互作用解析では c-fos/c-jun二量体との相互作用が知られているTPA-responsive element(TRE)の繰り返し配列を持つビオチン付きDNAをベイト,蛍光標識されたc-fos全長タンパク質,c-fosΔbZIP1タンパク質およびc-fos bZIPモチーフタンパク質の3サンプルとc-junタンパク質のヘテロ二量体をプレイとしている.
3.3 結果
3.3.1 プルダウン法を用いたタンパク質間相互作用の検証
プルダウン法を用いてc-fosタンパク質とc-fosΔbZIP1タンパク質のc-junタンパク質に対する相互作用能を比較したところ,互いにc-junのbZIPモチーフタンパク質との相互作用が確認された.しかし,相互作用強度には差があり,蛍光強度の相対値はベイトがc-fosの場合29.0であるのに対してc-fosΔbZIP1の場合22.0であった(図 3).
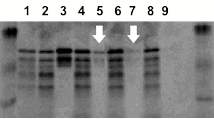
図 3:プルダウン法を用いたPPI解析の結果
1: input prey c-jun
bZIP motif, 2: Bait c-fos bZIP motif (supernatant), 3: Bait c-fos bZIP motif
(elution), 4: Bait c-fos (supernatant), 5: Bait c-fos (elution), 6: Bait
c-fosΔbZIP1 (supernatant), 7: Bait c-fosΔbZIP1 (elution), 8: No Bait (supernatant), 9:
No Bait (elution)
(分子量マーカー) BIO-RAD Kaleidoscope Restained Standards
161-0324
3.3.2
プルダウン法を用いたタンパク質-DNA相互作用の検証
プルダウン法を用いてc-fosタンパク質とc-fosΔbZIP1タンパク質のタンパク質-DNA相互作用能を比較すると,c-fosΔbZIP1は相互作用能をもたないことが確認された(図 4).従って.選択的スプライシングによってDNAとの相互作用能が失われ,さらにはc-fosがもつ転写因子としての機能を失うことが明らかになった.
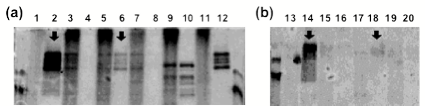
図 4:プルダウン法を用いたPDI解析の結果
(a) プルダウン反応液に翻訳阻害剤としてピューロマイシンを加えていない場合
1: Bait+
c-jun/c-fos (supernatant), 2: Bait+ c-jun/c-fos (elution), 3: Bait
DNA–
c-jun/c-fos (supernatant), 4: Bait– c-jun/c-fos (elution), 5: Bait+
c-jun/c-fosΔbZIP1
(supernatant) 6: Bait+ c-jun/c-fosΔbZIP1
(elution), 7: Bait– c-jun/c-fosΔbZIP1
(supernatant) 8: Bait– c-jun/c-fosΔbZIP1 (elution),
9: Bait+ c-jun/c-jun (supernatant)
10: Bait+ c-jun/c-jun (elution), 11: Bait+ c-jun/c-fos motif (supernatant), 12:
Bait+
c-jun/c-fos motif (elution)
(b) プルダウン反応液にピューロマイシンを加えた場合
13: Bait+ c-jun/c-fos (supernatant), 14: Bait+ c-jun/c-fos
(elution), 15: Bait DNA–
c-jun/c-fos (supernatant), 16: Bait– c-jun/c-fos (elution), 17: Bait+
c-jun/c-fosΔbZIP1
(supernatant) 18: Bait+ c-jun/c-fosΔbZIP1
(elution), 19: Bait– c-jun/c-fosΔbZIP1
(supernatant) 20: Bait– c-jun/c-fosΔbZIP1 (elution)
(Marker)
BIO-RAD Kaleidoscope Restained Standards 161-0324
3.4 考察
3.4.1 転写因子としてのc-fosΔbZIP1の機能変化
選択的スプライシングによってタンパク質間相互作用の強弱およびタンパク質-DNA相互作用の有無が確認された.以上より,c-fosΔbZIP1タンパク質がc-junタンパク質と結合し,二量体として機能する場合が考えられる.しかしc-fos/c-junがもつ強い転写活性能とは異なり,転写を抑制あるいは阻害する働きがあると予想される.また,この様に選択的スプライシングによって転写因子の機能が変化する例としてactivating transcription factor 3 (ATF3)のスプライスバリアントが報告されている(Hashimoto,
Y. et al. 2002).ATF3のスプライスバリアント(ATF3ΔZip2)は選択的スプライシングによってbZIPモチーフの一部を失う.この現象により,cAMP responsive element binding protein(CREB)との相互作用能は維持されるが,転写因子としての機能を失う.更に,HashimotoらはATF3ΔZip2が癌組織では確認されず,正常な細胞でのみ確認されていることから癌化との関連性があると考えている.
以上の様にFOSについてもATF3と同様に選択的スプライシングによる機能変化が生じている可能性がある.そこでATF3ΔZip2と同様にFOSΔbZIP1の組織特異性を確認することで表現型に及ぼす影響を調べた.
3.4.2 c-fosタンパク質の細胞内局在
c-fos/c-junの二量体が転写因子として働く場合,核内に移行する必要がある.この移行に必要な核移行シグナル(NLS)はc-fosタンパク質,c-junタンパク質共にもっているがc-junタンパク質のNLSに依存的であることが知られている(Roux,
P. et al. 1990).更にc-fosタンパク質の核移行シグナルは弱く,細胞質に局在することも知られている(Miller, D. et al. 1984).一方,本研究で用いたc-fosΔbZIP1は選択的スプライシングによってbasic
regionに存在するbipartite NLSを失う.つまり,c-fosΔbZIP1は細胞質において機能するタンパク質であることが示唆された.
次に,FOSおよび作成したFOSΔbZIP1について,green
fluorescent protein (GFP)との融合タンパク質を作製し,それらを用いて細胞内局在を調べたところc-fos/c-jun二量体はその大半が核内に局在することが確認された(図 5a).一方,c-fosΔbZIP1/c-jun二量体は多くが核内に移行するが,細胞質にも局在することが確認された(図 5b).
また,c-fos単独では核内に局在することが多く(図 5c),c-fosΔbZIP1単独では細胞質に局在することが多いという違いが確認された(図 5d).尚,本実験は慶應義塾大学先端生命科学研究所の小杉俊一講師に実施して頂いた.
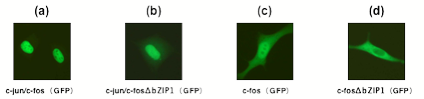
図 5:c-fosおとびc-fosΔbZIP1の細胞内局在
(a) c-fos/c-junの細胞内局在,
(b) c-fosΔbZIP1/c-junの細胞内局在,
(c) c-fosの細胞内局在,(d) c-fosΔbZIPの細胞内局在
以上より,選択的スプライシングは細胞内の局在に関係していることが明らかになった.従って,c-fosΔbZIP1はタンパク質間相互作用能が維持されているため,c-junが転写因子として働く際にその抑制因子あるいはインヒビターとしての機能をもつことが示唆された.
4 FOSスプライスバリアントの組織特異性検証
4.1 目的
選択的スプライシングと組織特異性との関連性については様々な研究がなされている.そこで,FOSΔbZIP1の組織特異性について検証を行い,その機能の推定を試みた.
尚,FOSΔbZIP1は選択的スプライシングデータベースのEASEDに登録されているスプライスバリアントの一つであり,膵臓および白血球由来のESTから予測されている.
一方で,FOSΔbZIP1の網羅的な組織特異性の情報は存在しない.そこで,複数の組織に由来するRNAやcDNAライブラリに対して,FOSΔbZIP1特異的なプライマーを用い, RT-PCR法で配列断片の増幅することで,FOSΔbZIP1の組織特異性の検証を行った.
4.2 対象と手法
4.2.1 6種の組織を用いたFOSΔbZIP1の検出
FOSΔbZIP1の組織特異性を確認するためにヒトの各組織から得られたmRNA(表4)を用いてRT-PCRを行った.尚,本実験では選択的エキソン周辺を増幅できるプライマーセットおよびmRNA全長を増幅できるプライマーセットを設計し利用した.また,この実験によってFOS全長mRNA由来の配列も増幅される.従って,FOS全長およびFOSΔbZIPの判定は電気泳動による泳動距離の差で検出することを試みた.
表 4:RT-PCRに使用した組織
|
Tissue |
Type |
Tissue |
Type |
|
Brain |
poly A+
RNA |
Liver |
poly A+
RNA |
|
Brain
Alzheimer |
poly A+
RNA |
Liver
Tumor |
poly A+
RNA |
|
Colon |
poly A+
RNA |
Pancreas |
Total RNA |
|
Colon
Tumor |
poly A+
RNA |
|
|
4.2.2
cDNAライブラリを用いたFOSΔbZIP1の検出
RT-PCRによる組織特異性の検証では網羅性が低いといえる.そこで,SpliceMiner cDNA Library Panel(STRATAGENE)という実験キットを用いた解析を行った.
この実験キットはヒトの癌組織28種類,正常組織32種類から得られた12,000個のcDNAクローンからランダムに384(96×4)のcDNAライブラリを作製したものである.
このキットを用いてPCR反応を行った.尚,プライマーセットにはFOS全長を増幅するものを使用しており,反応溶液にはPicoMaxx
High Fidelity PCR Master Mixを使用した.また,RT-PCRと同様に泳動距離の差によってFOSΔbZIP1を判定した.
4.3 結果
4.3.1 6種の組織を用いたFOSΔbZIP1の検出
RT-PCRによってFOS全長(全長配列:1,144bp,選択的エキソン周辺配列:330bp)は正常の脳組織,アルツハイマー病の脳組織,正常の大腸組織,腫瘍の大腸組織,腫瘍の肝臓組織でその発現が確認されたが,正常の膵臓組織と正常の肝臓組織では確認されなかった(図 6).一方,FOSΔbZIP1(全長配列:1,036bp,選択的エキソン周辺配列:222bp)は全ての組織で発現を確認できなかった.そこで,全長を増幅させるプライマーと選択的エキソン周辺を増幅させるプライマーの組み合わせを変えることでFOS全長との相違を確認したところ,3’側末端の配列が変化していることが確認された(図 7).
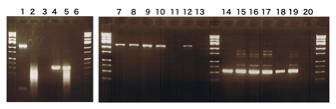
図 6:RT-PCRの結果
1: Brain (Full),2:Pancreas (Full),3:No mRNA (Full),4: Brain
(AE),
5:Pancreas (AE),6:No mRNA (AE),7:Brain (Full),8:Alzheimer (Full),9:Colon (Full),10:Colon Tumor (Full),11:Liver (Full),12:Liver Tumor (Full),13:No mRNA (Full),14:Brain (AE),15:Alzheimer (AE),16:Colon (AE),17:Colon Tumor (AE),18:Liver (AE),19:Liver Tumor (AE),20:No mRNA (AE),
Full:全長のプライマー,AE:選択的エキソンのプライマー
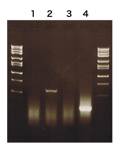
図 7:膵臓mRNAを用いたFOSバリアントの検証
1: 5’Full/3’Full,2:5’Full/3’AE,3:5’AE/3’Full,4:5’AE/3’AE
Full:全長のプライマー,AE:選択的エキソンのプライマー
4.3.2 cDNAライブラリを用いたFOSΔbZIP1の検出
SpliceMiner cDNA Library Panelを用いた実験の結果,FOSΔbZIP1(1,036bp)の発現が確認された(図 8).特に図 8のY64レーン,P33レーンでは高濃度のFOSΔbZIP1が確認された.
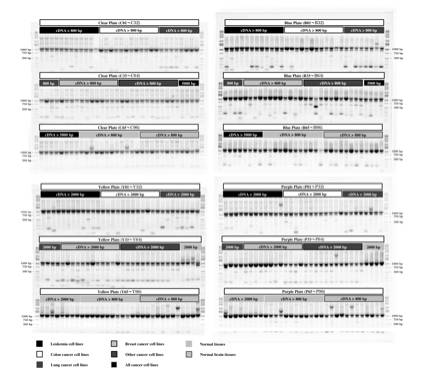
図 8:SpliceMiner cDNA
Library Panelを用いた実験結果
384サンプル(96×4サンプル)の泳動結果を示す.
尚,それぞれの色の枠はcDNAライブラリを作成した組織で分類されている.
また,枠内の数字はcDNAの配列長を示す.
4.4 考察
4.4.1 予測に利用したESTに対する実験結果
FOSΔbZIP1はEASEDでは膵臓のランゲルハンス島および白血球由来のESTによって予測されている.RT-PCRで用いた膵臓のtotal
RNAと比較すると,組織の部分と全体という相違がある.つまり,同一の組織でも部分的にスプライスパターンが異なる場合も考えられる.
また,疾患の有無も検討すべき点といえる.FOSは癌あるいは炎症性疾患の組織で高い遺伝子発現が認められており,癌のマーカー遺伝子としての利用も検討されている,今回はデータベースに疾患に関する情報がなかったため,実験では正常組織を使用したが,膵臓の疾患組織でも同様に検証することで新たな知見が得られるかもしれない.尚,図6のレーン11, 12は正常な肝臓細胞と腫瘍の肝臓細胞でFOSの転写物に顕著な差があることを示している.
4.4.2 RT-PCRとcDNAライブラリの比較から
RT-PCRではFOSΔbZIP1の転写物は確認されなかったため,その存在量は少ないと予想された.一方,cDNAライブラリを用いた組織特異性の確認によりFOSΔbZIP1が組織や疾患で特異的に発現している訳ではなく,多くの組織で発現していることが確認された.
更に,この結果からc-fosΔbZIP1に関する二つの事柄が推察された.
第一に,多くの組織で非特異的に発現していることからc-fosΔbZIP1が基本的な細胞機能の維持に関わっていることが考えられる.
第二に,FOSΔbZIP1の濃度が低い点については,c-fosの従来の働きへの影響が関わっていると考えられる.3.3.1で述べた様にc-fosΔbZIP1はc-junとの相互作用能を有する.また,3.3.2や3.4.2よりPDI,細胞内局在は変化する,仮にc-fosとc-fosΔbZIP1がほぼ等量存在していた場合,c-fosの従来の働きを阻害することになる.従って,c-fosΔbZIP1が機能する為にはc-fosの転写因子としての機能に影響を及ぼさない程度の低濃度で存在する必要があると考えられる.
4.4.3 FOSのスプライスバリアントの多様性
FOSスプライスバリアントは選択的スプライシングデータベースのASD Release2に6種類,ASAPに4種類,EASEDに13種類のスプライスバリアントが登録されている.今回のcDNAライブラリを用いた実験でもFOSΔbZIP1以外のバリアントも多く確認されている.特に図 8のB51レーンではおよそ500bpの配列がFOSよりも多く確認されており,癌組織(Other
cancer cell lines)由来である.また,図 8のY92レーンのNormal
brain tissueではおよそ2,000bpの配列の増幅が顕著である.
この様に,個々の組織で特異的に存在するスプライスバリアントとFOS全長やFOSΔbZIP1の様に組織によらず,常に転写が行われている様なスプライスバリアントが存在する.この違いによって制御に関わるもの,疾患に関わるものが存在するのではないかと考えられた.
5 結論
本研究はIVV法によって得られた網羅的な相互作用データから,選択的スプライシングによって相互作用能が変化する遺伝子候補を抽出する為の一連の解析工程を確立した.
更に,抽出された遺伝子候補から実際にFOSの選択的スプライシングによる相互作用能の変化について実験手法を用いて確認した.その結果,FOSは選択的スプライシングによってタンパク質間相互作用能は強弱の変化,タンパク質-DNA相互作用はON/OFFの変化が引き起こされ,核内と細胞質という様に細胞内の局在パターンを変化することも確認された.
また,組織特異性の検証をおこなった結果,多くの組織で確認され,一部の組織で存在量が多かったことから,FOSスプライスバリアントの翻訳産物であるc-fosΔbZIP1がc-fosとの存在量を適切に設定し機能していることが予想された.
以上より,タンパク質間相互作用領域の情報と遺伝子情報の変化を組み合わせてタンパク質機能の変化を推定する手法を構築できたといえる.今後,FOS以外に抽出された遺伝子についても同様に検討を行うことで,選択的スプライシングという生命現象の本質に迫ることができると思われる.
謝辞
本研究を行うにあたり,慶應義塾大学理工学部柳川研究室の宮本悦子講師,藤森茂雄氏,石坂正道氏,同大学政策・メディア研究科の北川統之氏,万有製薬株式会社の鷲尾尊規氏には有用な助言を頂きました.また実験に際しては政策・メディア研究科の梅木瞳氏,理工学部柳川研究室の平井直也氏,増岡和代氏には大変お世話になりました.更に,慶應義塾大学先端生命科学研究所の小杉俊一講師には有益な実験結果を提供頂きました.この場を借りて厚く御礼申し上げます.最後にこのような研究の機会を与えて下さった慶應義塾大学理工学部柳川弘志教授,同大学環境情学部冨田勝教授には深い感謝の意を表したいと思います.
参考文献
Altschul, S.F., Madden, T.L., et al. (1997). "Gapped BLAST and
PSI-BLAST: a new generation of protein
database search programs." Nucleic Acids Res 25(17): 3389-402.
Hashimoto,
Y., Zhang, C., et al. (2002). “An alternatively spliced isoform of transcriptional
repressor ATF3 and its induction by stress stimuli.” Nucleic Acids Res 30(11): 2398-406.
Kan, Z., States, D., et al. (2002). "Selecting for functional
alternative splices in ESTs."
Genome Res 12(12): 1837-45.
Lee, C., Atanelov, L., et al. (2003). "ASAP: the Alternative
Splicing Annotation Project."
Nucleic Acids Res 31(1): 101-5.
Miller, A.D.,
Curran, T., et al. (1984). “c-fos protein can induce cellular
transformation: a novel mechanism of activation of a cellular oncogene.” Cell 36(1): 51-60.
Mironov, A.A., Fickett, J.W., et al. (1999). "Frequent alternative
splicing of human genes." Genome Res 9(12): 1288-93.
Miyamoto-Sato, E., Ishizaka, M., et al. (2005). "Cell-free cotranslation and
selection using in vitro
virus for high-throughput analysis of
protein-protein interactions and complexes." Genome Res 15(5):
710-7.
Modrek, B. and Lee, C. (2002). "A
genomic view of alternative splicing." Nat Genet 30(1): 13-9.
Modrek, B., Resch, A., et al. (2001). "Genome-wide detection of
alternative splicing in expressed sequences of human genes." Nucleic Acids
Res 29(13): 2850-9.
Pospisil, H., Herrmann, A., et al. (2004). "EASED: Extended
Alternatively Spliced EST Database." Nucleic Acids Res 32(Database issue):
D70-4.
Roux, P.,
Blanchard, J.M., et al. (1990). “Nuclear localization of c-Fos, but not
v-Fos proteins, is controlled by extracellular signals.” Cell 63(2): 341-51.
Thanaraj, T.A., Stamm, S., et al. (2004). "ASD: the Alternative
Splicing Database." Nucleic Acids Res 32(Database issue): D64-9.
Xing, Y., Xu, Q., et al. (2003). "Widespread production of
novel soluble protein isoforms by alternative splicing removal of
transmembrane anchoring domains."
FEBS Lett 555(3): 572-8.
Xu, Q., Modrek, B., et al. (2002). "Genome-wide detection of
tissue-specific alternative splicing in the human transcriptome." Nucleic
Acids Res 30(17): 3754-66.