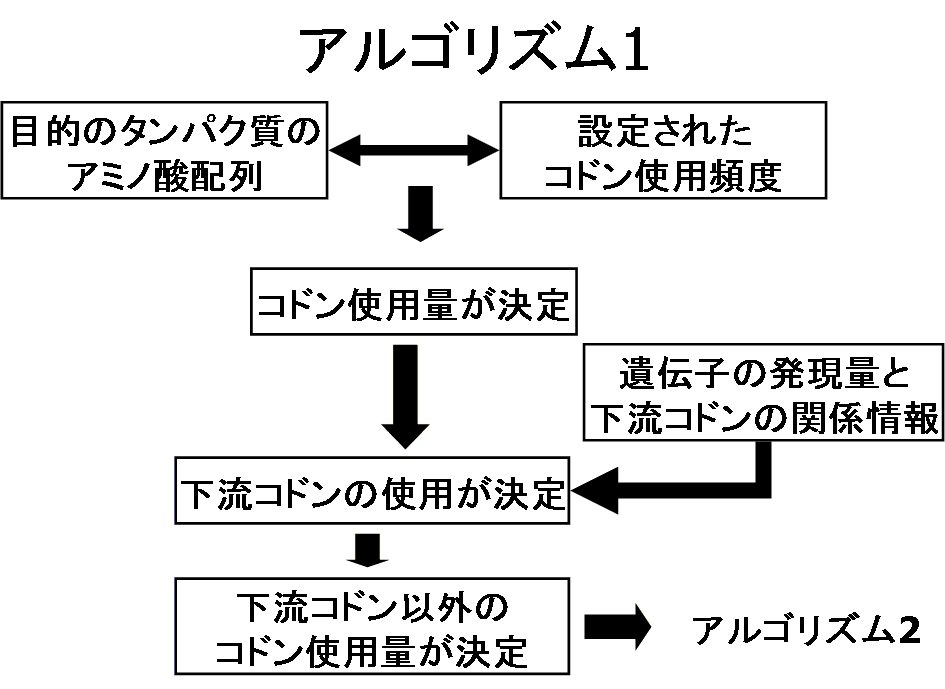
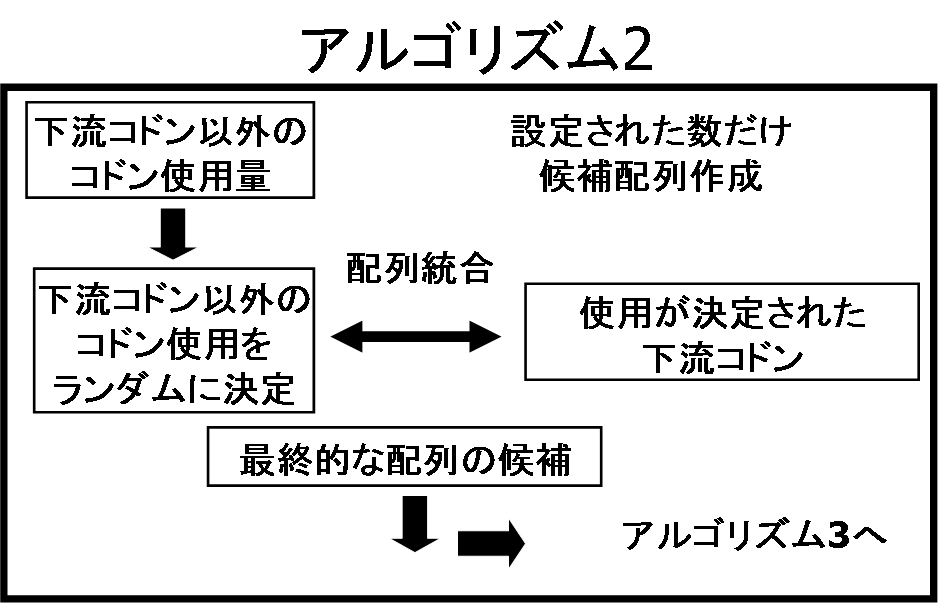
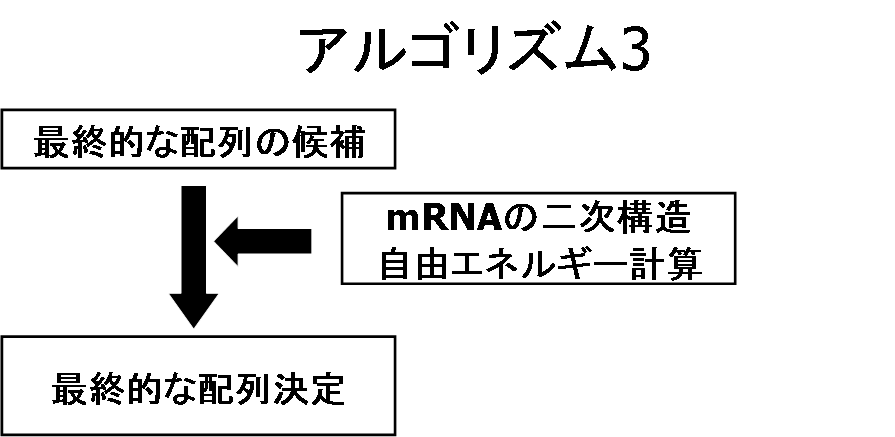
 図1 ASOSによるタンパク質高発現に適したDNA配列設計の概念図
図1 ASOSによるタンパク質高発現に適したDNA配列設計の概念図
|
4. 「結果」
枯草菌を宿主としたタンパク質高発現に適したDNA配列を選択するソフトウェアであるASOSを改良する事で,20種類以上の微生物(Borrelia burgdorferi,Burkholderia pseudomallei,Clostridium perfringens,Chlamydia trachomatis,Escherichia coli,Helicobacter pylori,Hemophilus influenzae,Mycobacterium tuberculosis,Staphylococcus aureus,Streptococcus pneumoniae,etc)においてタンパク質高発現に適したDNA配列を選択できるようにした.
5. 「今後の展望」
今後は,ASOSの有用性の実験的な検証を行なったうえで,一般のユーザー向けに本ソフトウェアの公開を行いたいと考えている.
6. 「参考文献」
[1] SHIMOMURA, O., JOHNSON, FH. and SAIGA, Y. (1962) Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. J Cell Comp Physiol, 59, 223-239
[2] http://en.wikipedia.org/wiki/Green_fluorescent_protein
[3] Ayoub, NA., Garb, JE., Tinghitella, RM., Collin, MA and Hayashi, CY. (2007) Blueprint for a high-performance biomaterial: full-length spider dragline silk genes. PLoS ONE. 13,514
[4] Kanekiyo, T., Ban, T., Aritake, K., Huang, ZL., Qu, WM., Okazaki, I., Mohri, I., Murayama, S., Ozono, K., Taniike, M,. Goto, Y. and Urade, Y. (2007) Lipocalin-type prostaglandin D synthase/beta-trace is a major amyloid beta-chaperone in human cerebrospinal fluid. Proc Natl Acad Sci U S A. 104,6412-6417
[5] タンパク質発現受託のトータルサービスー-遺伝子合成から抗体作成まで- タカラバイオ株式会社遺伝子工学研究製品・受託サービス情報
[6] 糖谷大樹 (2003) 組み換えタンパク質発現系の特徴と問題点 東レリサーチセンター THE TRC NEWS No.82
[7] 米田俊浩 近藤恵二 (1996) 構造解析のための蛋白質作成技術-酵母を用いた蛋白質の生産- 構造生物 Vol.2 No.2
[8] Huemmerich, D., Helsen, CW., Quedzuweit, S., Oschmann, J., Rudolph, R. and Scheibel, T. (2004) Primary structure elements of spider dragline silks and their contribution to protein solubility. Biochemistry. 43,13604-13612
[9] Ohashi, Y., Yamashiro, A., Washio, T., Ishii, N., Ohshima, H., Michishita, T., Tomita, M. and Itaya, M. (2005) In silico diagnosis of inherently inhibited gene expression focusing on initial codon combinations. 347, 11-9
[10] Kanaya, S., Kudo, Y., Nakamura, Y. and Ikemura, T. (1996) Detection of genes in Escherichia coli sequences determined by genome projects and prediction of protein production levels, based on multivariate diversity in codon usage. Comput Appl Biosci. 12, 213-225
[11] Kubo, M. and Imanaka, T. (1989) mRNA secondary structure in an open reading frame reduces translation efficiency in Bacillus subtilis. J Bacteriol 171,4080-4082
[12] 中津大貴.(2008) 慶應義塾大学環境情報学部 卒業論文
[13] Ohashi, Y., Yamashiro, A., Washio, T., Ishii, N., Ohshima, H., Michishita, T., Tomita, M. and Itaya, M. (2005) In silico diagnosis of inherently inhibited gene expression focusing on initial codon combinations. 347, 11-9
[14] http://www.ncbi.nlm.nih.gov/
[15] http://au.expasy.org/sprot/


 図1 ASOSによるタンパク質高発現に適したDNA配列設計の概念図
図1 ASOSによるタンパク質高発現に適したDNA配列設計の概念図


 図1 ASOSによるタンパク質高発現に適したDNA配列設計の概念図
図1 ASOSによるタンパク質高発現に適したDNA配列設計の概念図