要旨
In vitroタンパク質キナーゼがリン酸化を行う際に標的とする残基周辺には,「モチーフ配列」と呼ばれる特徴配列が存在する.この様な各キナーゼ特異的な基質の配列条件は,基質情報を基にした上流キナーゼの予測に利用できる.本研究では,キナーゼ特異的なモチーフ配列を同定する為に,in vitroキナーゼ法を基にした実験手法を構築した.細胞破砕物由来の脱リン酸化タンパク質混合試料をキナーゼと反応させ,リン酸化部位を液体クロマトグラフィー-タンデム質量分析 (LC-MS/MS) によって定量的に解析することで,あるキナーゼ特異的なリン酸化部位を同定した.このリン酸化部位周辺配列からモチーフ配列を抽出することにより,キナーゼ-基質間にみられる認識関係を明らかにした.
序論
近年,リン酸化反応を行うキナーゼの同定が進み,ヒトは500以上のタンパク質キナーゼを持つ事が明らかになった (Manning, et al., 2002).ところが,リン酸化反応が媒介するシグナル伝達経路はキナーゼのみならず,それらが標的とする基質を含む多様な分子が相互に関わり合う事で構成されている.シグナル伝達経路の詳細な構造を理解するためには,キナーゼと基質の双方に関して理解を深める必要がある.また,大量のリン酸化部位が同定されている一方で,その大部分は既知のシグナル伝達経路上にマップされていない.LC?MS/MSによる解析結果からは,リン酸化を行った上流キナーゼを同定することができず,どのようにシグナル伝達に関わるのか明らかとなっていない基質が多く存在する.これらの問題を解決するために,キナーゼの基質特異性が注目されている.
各キナーゼは基質リン酸化部位周辺に位置する特徴的なアミノ酸配列である「モチーフ配列」を認識することが知られている (Kobe, et al., 2005).これらのモチーフ情報をリン酸化情報からキナーゼを特定するためのルールとして利用する事により,LC-MS/MSによって大量同定された基質情報からキナーゼを探索する際の足掛かりとなる.これまで,既存の実験的手法に加え,新たな情報学的手法を応用する事で多くのキナーゼとそれらが認識する基質のモチーフ配列が検出され,HPRD (Human Protein Reference Database) (Keshava Prasad, et al., 2009),PhosphoSitePlus (Cell Signalings Technology),また NetPhorest (Miller, et al., 2008) などの様々なデータベースに登録されている.ところが,モチーフ配列情報が明らかとなっているキナーゼの数は少ない.入手可能な情報が未だに欠損しており,モチーフ情報を基に基質のキナーゼを予測するNetPhorestには,既知のヒトキナーゼ518 (Manning, et al., 2002) のうち,約35%に対応するモチーフ配列しか登録されていない.また,モチーフ配列を得るために根本的に必要とされるキナーゼ-基質ペア情報も依然として欠如している.例えば,生体内のみならず試験管内で実験的に検証されたおよそ22,000事例のリン酸化部位情報を含有する公共データベースであるPhospho.ELMでは,対応するキナーゼが判明している事例はわずか21%である (Diella, et al., 2008).この様に,キナーゼ?基質情報およびモチーフ配列の情報不足は,シグナル伝達経路および大規模リン酸化ネットワークの解明を限定している.
これらの問題の一因として,キナーゼ?基質ペアを大規模に同定するための実験的手法がなかった事があげられる.そこで,より簡便かつ汎用性の高い実験法を構築する為に,本研究ではin vitroキナーゼ反応を利用し,これらの問題の解決し,モチーフ配列の特定を試みた.本手法では,脱リン酸化反応を伴うin vitroキナーゼ反応と,本研究室で開発されたリン酸化ペプチド濃縮法であるHAMMOC法 (Sugiyama, et al., 2007) を用いたLC-MS/MSによるリン酸化部位の網羅的な検出手法を合わせる事で,複数のキナーゼに応用可能,且つ比較的短時間で実行可能な実験手法を構築し,少量タンパク質からキナーゼ特異的な大量のリン酸化部位同定を実現した.
結果と議論
1. 実験手法の構築
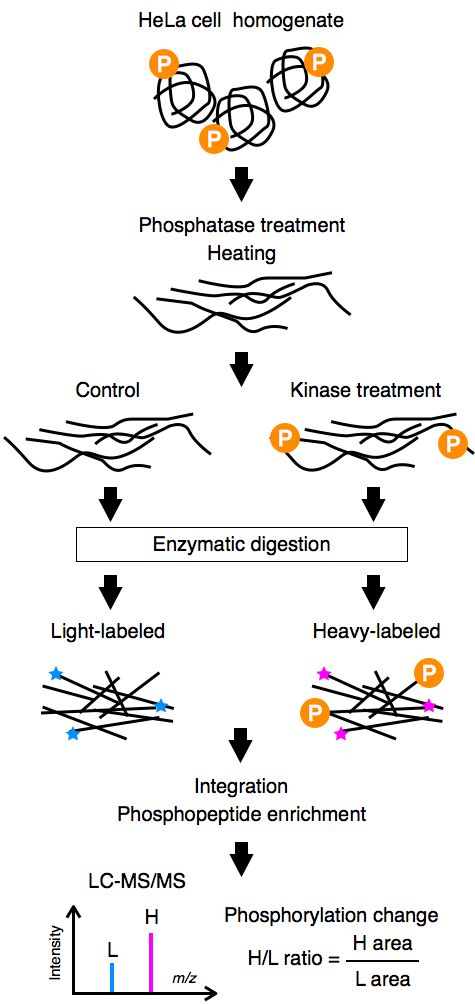
本実験手法ではヒト培養細胞であるHeLa細胞を破砕し,タンパク質を抽出した.アルカリ脱リン酸化酵素 (AP; alkaline phosphatase) を用いて,細胞内にて付与されたリン酸基を取り除いた後に,加熱処理を行い,これによりAPや内在性キナーゼを不活性化した.ヒトのリコンビナントキナーゼでリン酸化を行ったタンパク質を,2種類のエンドプロテイナーゼを用いてペプチドレベルに消化し,リン酸化ペプチドのみを選択的に濃縮した後に,LC-MS/MS解析を行いてキナーゼによるリン酸化部位を大規模同定した.
脱リン酸化タンパク質に対してin vitroキナーゼ法とLC-MS/MSを用いて基質同定を行うという上述の実験法は,マウスの子宮組織内のタンパク質プール内で,cAMP依存性プロテインキナーゼ (PKA; cAMP-dependent protein kinase) またはcGMP依存性プロテインキナーゼ (PKG; cGMP-dependent protein kinase) が標的とする基質リン酸化部位の探索に利用されたという報告がある (Huang, et al., 2007).ところが,この先行研究により同定されたin vitroでの基質タンパク質は,PKAおよびPKGに対してそれぞれで61または12と少なかった.
そこで,この手法に2点の変更を加える事で,キナーゼ特異的なリン酸化部位の大量同定をより効果的に行う為のin vitro実験系を構築した (図参照).具体的な変更点は,脱リン酸化反応に用いる酵素の選抜と,安定同位体標識法の導入である.第一に,より効率の良い脱リン酸化酵素を用いてタンパク質のリン酸基を取り除く事で,細胞内のリン酸化状態がキナーゼ反応結果に偏りを及ぼさない様にする.なるべく多くのリン酸基をキナーゼ反応の前に取り除く事で,キナーゼにより微量にリン酸化されるような部位も検出できる様になる.第二に,安定同位体標識法による相対定量を行う事で,コントロールとの比較を行い,脱リン酸化後、あるいはリン酸化後のタンパク質がどの程度リン酸化を受けたのか数量的に示した.また,この実験系に当研究室内で開発された効果的なリン酸化ペプチド濃縮法 HAMMOC法 (Sugiyama, et al., 2007) を組み合わせる事で,より大量かつ精密なキナーゼ特異的なリン酸化部位の同定が可能となった.
このハイスループットな実験系を用いて解析基盤として充分な量のリン酸化部位情報を獲得した後,情報解析手法を用いてもチーフ配列を特定した.
2. In vitroリン酸化部位情報を用いたモチーフ配列の抽出
 In vitroでの反応系は,in vivoの様な生体内反応をそのまま反映するとは限らない.その半面,物質の物理化学的性質に依存して反応が起きることが期待される.タンパク質キナーゼは,その基質中の特徴的な配列を認識してリン酸化を行う事が知られている.そのため,in vitroにてキナーゼが行うリン酸化反応を網羅的に検出することにより,キナーゼがその性質特異的に好むモチーフ配列を検出できると考えた.そこで,In vitroキナーゼ反応とLC-MS/MSを組み合わせた実験手法により (結果1),ある特定のキナーゼによりおこるリン酸化反応を大規模かつアミノ酸部位レベルで特定し,モチーフ配列の抽出を試みた.
In vitroでの反応系は,in vivoの様な生体内反応をそのまま反映するとは限らない.その半面,物質の物理化学的性質に依存して反応が起きることが期待される.タンパク質キナーゼは,その基質中の特徴的な配列を認識してリン酸化を行う事が知られている.そのため,in vitroにてキナーゼが行うリン酸化反応を網羅的に検出することにより,キナーゼがその性質特異的に好むモチーフ配列を検出できると考えた.そこで,In vitroキナーゼ反応とLC-MS/MSを組み合わせた実験手法により (結果1),ある特定のキナーゼによりおこるリン酸化反応を大規模かつアミノ酸部位レベルで特定し,モチーフ配列の抽出を試みた.
今回の試行実験には,セリン・スレオニンキナーゼである細胞外シグナル制御キナーゼ (ERK1; extracellular-signal-regulated kinases 1),およびPKAを用いた.これらのキナーゼを利用した理由は,比較的多数の基質部位が検出されやすい事,すでに研究が盛んに行われており得られた基質部位と照合する為の正解セットが多く存在する事,さらにERK1とPKAの既知モチーフ配列は傾向が異なる事があった.ERK1はリン酸化部位の周辺にプロリン (P; proline) を持つモチーフが多く,一部のモチーフはリン酸化部位の前またはN末端側にK (K; lysine) を持つものが知られている.また,PKAのモチーフ配列では,リン酸化部位のC末側にKやアルギニン (R; arginine) の様な塩基性の残基がよく出現する.ERK1によるリン酸化反応の結果,全部で473カ所のリン酸化部位が同定され,その内pSは347カ所,pTは121カ所,またpYは4カ所同定された.また,PKAによるリン酸化反応の結果,935カ所のリン酸化部位が同定され,pS,pTおよびpYはそれぞれ855カ所,72カ所,7カ所が検出された.これらのリン酸化部位に対して,既存のモチーフ抽出ソフトウェアであるmotif-x (Schwartz and Gygi, 2005) を用いてモチーフ配列の探索を行った.
その結果,ERK1のリン酸化部位周辺配列からは,Pが-2または+1に含まれるPxpSP,pSP,pTPといった,既知モチーフと類似したモチーフ配列が数種類抽出された (上図A).また,pSAというデータベースに登録のない配列も検出された.その一方で,既知モチーフとして知られている,-1の位置にKを持つようなモチーフ配列は抽出されなかった.このことから,Kはキナーゼに対する親和性が比較的低く,ERK1が標的基質を決めるためにはPの存在が重要である可能性が示唆された.PKAのリン酸化部位 (上図B) からは,C末側にRやKをもつ配列が複数抽出された (RxxpS,RxpS,RxxxpS,KxxpS,KxpS,RxxxxxpS,RxxxxpS,RxxpT).この特徴は既知のモチーフ配列と一致している.また,PKA既知モチーフ配列と一致しない結果として,GpSというモチーフ配列が見つかった.
PhosphositePlusに登録のあるERK既知基質のうち,リン酸化部位の直後にAが存在し,また周辺にPを持たない基質リン酸化部位は3つ存在した.その内,ヒトのin vivo実験にてERK1/2によるリン酸化が確認されたPXN (Paxillin) は複数のドメインを持つアダプタータンパク質であり,多数のキナーゼからリン酸化を受ける.しかし,EGFRとPKCを媒介とする細胞増殖の系において,ERK1/2によるPXNのS126へのリン酸化修飾が必要であると確認されている (Sen, et al., 2010).また一方で,Kv4.2のS552に対しても,ヒトのin vivo実験にてPKAによるリン酸化が確認されている.Kv4.2は電位依存性カリウム (Kv, Voltage-gated potassium) チャネルの一種で,神経の興奮性制御にかかわっている.PKAによるKv4.2のS552へのリン酸化は,その特異的なKChIP (Kv channel interacting protein) との相互作用を通したKv4.2の輸送において,重要な役割を果たしている (Lin, 2010).pSAやGpSといった配列を持つアミノ酸へのリン酸化が,非特異的な反応ではなく,生体機能に関わるという事により,今回検出された配列がキナーゼの基質認識に利用されている可能性を示した.しかし,多数のリン酸化部位がこのモチーフ配列候補とマッチする訳ではなく,このモチーフがin vitro環境特異的に起こるリン酸化反応の影響である可能性を完全には否定できない.モチーフ配列として確証を得るには,基質モチーフ配列に変異を入れた際のリン酸化量変化や,in vivoでの上流キナーゼ阻害によるリン酸化量変化など,更なる検証の余地がある.
3.結論
In vitroキナーゼ法とLC-MS/MSを組み合わせた実験法を構築し,キナーゼ特異的な基質リン酸化部位を大量同定した.本手法では,細胞を破砕物からタンパク質を取得し,脱リン酸化反応を行う.脱リン酸化タンパク質に対して,ERK1またはPKAによるリン酸化反応を行い,その後コントロールサンプルおよびリン酸化タンパク質に対して安定同位体標識を行う事により,LC-MS/MSにてキナーゼ反応によるリン酸化の定量的な測定を可能とした.In vitroキナーゼ反応によりリン酸化が増大していた残基の周辺配列情報を取得し,モチーフ探索に利用した.その結果,ERK1とPKAそれぞれに特異的なモチーフ配列が抽出された.このことから,今回構築した実験系はキナーゼ特異的なモチーフ配列を同定するために利用できることが示唆された.本手法は簡便かつ単純であるため,その他のキナーゼへ適用する事で網羅的なモチーフ配列比較に応用できる.
今年度の実績
1. 論文採択
- "Towards the systematic discovery of signal transduction networks using phosphorylation dynamics data"
- BMC Bioinformatics, BioMed Central Ltd, 11:232, 2010.
- Haruna Imamura, Nozomu Yachie, Rintaro Saito, Yasushi Ishihama, Masaru Tomita
2. 学会発表 (ポスターおよび口頭発表)
- "In vitro実験系による網羅的なキナーゼ/基質情報の探索に向けて"
- 第33回日本分子生物学会年会・第83回日本生化学会大会 合同大会・兵庫, 神戸, 2010/12/07-10
- 今村春菜, 杉山直幸, 冨田勝, 石濱泰