
ネットワークの構築とマイニング 慶應義塾大学先端生命科学研究所・環境情報学部
斎藤輪太郎
本研究では、コンピュータを使ってモデル生物である大腸菌の信頼性の高いタン パク質間相互作用ネットワークを構築し、そこからの生物学的知見の発見を目指 している。タンパク質間相互作用は複数のタンパク同士が結合する現象であり、 DNA情報の転写、タンパク質合成、DNAの複製、シグナル伝達など実に多くの生命 現象に関わっている。現在酵母菌などいくつかの種において、実験で得られた多 量のタンパク質間相互作用データが入手可能であり、これらのデータの中には様々 な新規生物学的情報が詰まっているはずである。しかし実験で得られたタンパク 質間相互作用データの半分はエラーと見積もられており、これらの貴重な情報の 発見を非常に困難にしている。一方で、相互作用するタンパク質のペアは遺伝子 発現や系統プロファイル上の相関があることが示されている。そこで大腸菌を用 い、これらのデータとタンパク質間相互作用の実験データを統合し、実験データ の信頼性の重み付けや、新規タンパク質間相互作用の予測を行った。

同時にPull-downの実験より得られたタンパク質間相互作用データを用意した(※)。 そしてPositive PPIとNegative PPIで(1)遺伝子の発現相関、(2)系統プロファイ ル上の相関、(3)Essentialな遺伝子か、(4)どのようなタンパク質のモチーフの 組み合わせを持っているか、 (5)実験の再現性がどの程度あるか、解析した。そ の結果、Positive PPIの方がNegative PPIに比べ(1)と(2)が有意に高く、また (3)Essentialな遺伝子を含む可能性が高かった。(4)に関しては実験データより 有意に出現するモチーフの組み合わせを評価する指標MMI scoreを開発した。(5) に関しては、実験の再現回数およびネットワークのトポロジーより実験の再現性 を評価する指標IGを開発した。その結果、Positive PPIの中ではNegative PPIに 比べ、(4)MMI scoreも(5)IGも有意に高いことが示された。

そこでこれら(1)〜(5) の特徴をもとに、相互作用をする可能性が高いタンパク質の組み合わせを抽出す るアルゴリズムを開発した。具体的には(1)〜(5)の指標をベイズ統計の枠組みを 使って統合し、対数オッズ比と呼ばれる1つのスコアにした。このスコアが高け れば高いほど、与えられたタンパク質のペアは相互作用する可能性が高いことを 意味する。これを用いて大腸菌の全てのタンパク質の組み合わせを評価し、スコ アが高いペアの抽出を行った。
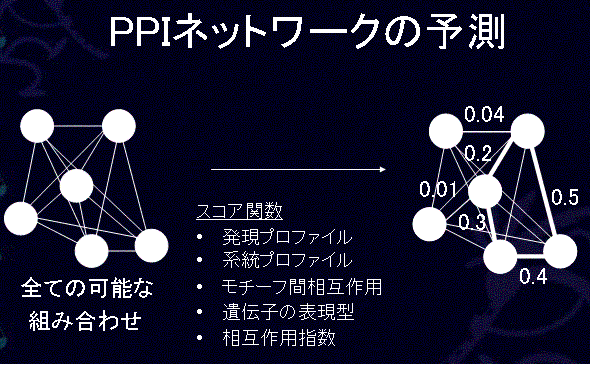
こうして予測された相互作 用の集合には既知の相互作用が、ランダムデータに比べ、250倍以上濃縮されて いることが判明し、また実験データと比較しても2倍程度濃縮されていることが 示された。
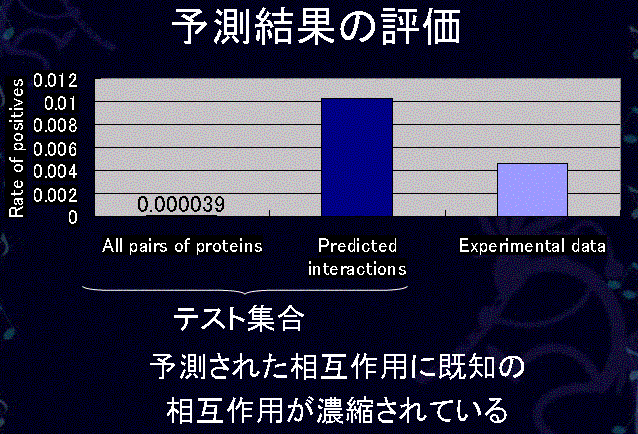
このように、タンパク質間相互作用に関わる様々な指標を統合し、ゲノムワイド にタンパク質間相互作用を高い精度で予測することに成功した。
※実験データは全て奈良先端科学技術大学院大学森研究室および京都大学和田研究室より提供して頂いた。