2012年度森基金研究成果報告書
研究課題名
大腸菌機能未知酵素YhhYのタンパクアセチル化能の有無の検証
慶應義塾大学 政策・メディア研究科
井内 仁志
概要
酵素は生物の代謝を担う重要なタンパク質であり,古くから新規酵素の機能同定は生物学の主要な分野の一つだった.しかし,最もよく実験室で用いられている大腸菌でさえ全酵素のうち約40%はその機能が実験的に証明されていない.このことは代謝研究を行う上で問題となっており,迅速に代謝経路上の穴を埋めることが望まれている.そこで本研究では,質量分析計を用いて大腸菌の機能未知酵素
YhhY の機能解明を目指した.まず,YhhY,20種のアミノ酸とアセチル CoA を一晩反応させ反応液を質量分析計で測定した.その結果,YhhY がヒスチジン,メチオニンとフェニルアラニンをアセチル化することが明らかになった.さらに,YhhY の生体内での働きを明らかにするため,yhhY 遺伝子欠損株のメタボローム解析を行った.その結果,野生型からも変異型からも 3 種のアセチル化アミノ酸は検出されず,さらにメタボロームに大きな差は無かった.そこで,yhhY 遺伝子を過剰発現させた株とさせない株のメタボローム解析を行ったところ,yhhY遺伝子を過剰発現した株でのみ3種のアセチル化アミノ酸の蓄積が確認された.これらの結果から,YhhY は生体内でもアミノ酸のアセチル化に関わっていると考えられる.今期は更に高濃度コバルト存在下でのメタボローム解析を行った.その結果,高濃度コバルト存在下においてYhhYは鉄-硫黄クラスターに関連して重要な役割を担っている可能性が示唆された.このように本研究によって YhhY の生体内外での働きが明らかになった.
キーワード: Eschserichia coli, yhhy, キャピラリー電気泳動-質量分析計, metabolome
背景
酵素は生物の代謝を担う重要なタンパク質であり,古くから新規酵素の機能同定は生物学の主要な分野の一つだった.しかし,最もよく実験室で用いられている大腸菌でさえ全酵素のうち約40%はその機能が実験的に証明されていない (Chen and Vitkup, 2007).同様に,大腸菌の遺伝子,タンパク質,代謝経路のデータベースであるEcoCycでは30-40%の代謝経路は,存在が確認されているにも関わらずその反応を担っている酵素が割り当てられていない『Orphan activity』であるとしている(Keseler et al., 2005).現時点の生命科学においてこれらの代謝地図上の欠落は代謝研究を行う上で大きな障壁であり,迅速にこれらの欠落を埋めることが望まれている.
ここで,近年の質量分析計の測定精度の向上はタンパクの機能同定に大きく貢献している.Saghatelianらは野生型と脂肪酸合成系の酵素を欠損したマウス組織のメタボロームを比較することで基質を同定している(Saghatelian et al., 2005).彼らが用いた手法は生体の組織メタボロームを測定することで基質を直接同定できる優れた手法である.しかし,明らかにしたい経路が1)可逆反応,2)アイソザイム,3)バイパス経路を持つ場合,遺伝子欠損が必ずしもその基質の蓄積に影響を与えるとは限らない.よって,そのような条件下では酵素機能の同定が出来ない可能性が高く,汎用的な手法でない.そこで,Saitoらはこれらの問題を解決したMetabolic Enzyme and Reaction discovery by Metabolite
profile Analysis and reactant IDentification
(MERMAID) 法を確立した.MERMAID法では酵母抽出物を溶解した『メタボライトジュース』の中に精製した大腸菌のタンパクを加えて反応させ,その反応液とメタボライトジュースのメタボロームをCapillary Electrophoresis-mass spectrometry (CE-MS) で分析する.減少した物質はその酵素が触媒する反応の基質であり,増加した物質はその反応の生成物である.筆者らはMERMAID法によって大腸菌のYihUがNADH依存的にコハク酸セミアルデヒドをガンマヒドロキシ酪酸に還元する働きを持つことを明らかにした (Saito et al., 2006, 2009).さらに,Carvalhoらも同様の手法で他の微生物の酵素機能同定を行なっており,Mycobacterium tuberculosisのRV1248cタンパクとMycobacterium bovisの抽出液を反応させ生成物をLiquid Chromatography-mass spectrometry (LC-MS) で測定し,RV1248cが2-ヒドロキシ-3-オキソアジピン酸生合成酵素だと同定した(Carvalho et al., 2010).
しかし,MERMAID法は基質と生成物の定量に時間がかかるのが欠点であった.なぜなら,これらの手法は酵素と数千の代謝物を含む生物の抽出液を反応させるため,質量分析の前にキャピラリー電気泳動や液相クロマトグラフィーで代謝物を分離する必要があり,このステップに多大な時間がかかるためである.よって,これらの手法によってハイスループットに機能同定を進めるのは難しかった.モデル生物であり,分子生物学において最もよく研究されてきた大腸菌ですら全体の約40%の酵素の機能がわかっていないことを鑑みると,1サンプルにつき数分で測定できる手法が必要とされている.
ここで,近年の大腸菌に関するデータベースの充実は機能未知酵素の基質候補を絞り込むことを可能にした.多くの酵素は情報学的に予測された基質候補がデータベース上で公開されていおり,その数は急速に増加している.
そこで,本研究ではデータベースで公開されている情報を用いて質量分析計による酵素機能同定法の高速化を目指した.酵素とデータベースで公開されている数十種の基質候補のみで反応させれば,試験管内にはごく限られた代謝物質しか存在しない.よって,質量数のみで物質同定できるのでCEやLCで分離する必要がなくなる.そのため,数千の代謝物質を含む『メタボライトジュース』を基質として用いる方法よりも測定時間が大幅に短縮出来るようになる.測定時間の短縮は機能同定効率の向上を可能にする.エラー! 参照元が見つかりません。に本手法の概略を示した.まず,Kyoto
Encyclopedia of Genes and Genomes (KEGG) など公共データベースから,まだ機能を実験的に証明されていない酵素を抽出する.次に,その酵素の遺伝子をプラスミドに持った大腸菌株をASKA libraryから取得し,その酵素を精製する (kitagawa et al.,
2005).精製した酵素と予想される基質を試験管内で反応させる.このとき,酵素を加えていない対照群も用意する.これはその反応が確かに加えた酵素によって起きているのかどうかを確認するためである.最後に,質量分析計で生産物が合成されていることを確認する.反応が進んでいれば基質が減少し,生成物が検出されるはずである.
ここで,情報学的にその機能が予測されている酵素についても実験的証拠を得ることは非常に重要である.例えば,AlexandraらはKEGGで公開されている6つの酵素群毎の”misannotation”率を調べたところ,ある酵素群では63%の酵素が間違ったアノテーションを付けられていることを報告した(Alexandra et al., 2009).このように,生命情報学によるアノテーションはアミノ酸配列から得られた『予測』であり,その酵素の持つ本来の機能と異なる場合も多い.よって,アノテーションが付いている酵素についてもその機能実験的に証明し,生化学的な知識を積み重ねることは,ポストゲノム時代においても重要な仕事である.
今回は手法確立のためのモデルケースとして大腸菌の機能未知酵素YhhYを用いた,YhhYはアセチル基転移酵素としての機能を持つと考えられているが,その実験的な証拠はまだない. なお,本タームペーパーは論文のドラフトとして作成しているために先学期までに行った実験の結果も含めている.
対象と手法
使用した菌株と培養条件
タンパク精製用及び過剰発現株のメタボローム測定用のyhhY遺伝子過剰発現株はASKA cloneから取得した (Kitagawa et al., 2005) .コントロール株は環状プラスミドの入っていないAG1株にプラスミドpCA24Nを挿入したものを用いた.欠損株はKeio CollectionからyhhY遺伝子を欠損したJW3504株とコントロール株としてBW25113株を使用した (Baba et al., 2006).メタボロームサンプリング用の培養は500mlバッフル付きフラスコでバッチ培養によって行った.菌株のグリセロールストックを1.5%LB寒天培地 (1L中に10g tryptone,5g east extract,10g NaCl)
に撒きコロニー化させ,楊枝でコロニーを拾いyhhY過剰発現株は2ml LB培地に,yhhY欠損株は2ml M9培地 (Wanner et al., 2001) に植菌した(n=3).対数増殖期が終わったら50ml の培地が入ったフラスコに一晩前培養した培養液をOD600 0.05になるように植菌し37℃,120rpmで培養した.
酵素精製
酵素の過剰発現はOvernight Express Autoinduction system (Merk社) を用いた.操作はキット付属の説明書に従った.LB培地で前培養した大腸菌を説明書通りに溶液を加えた1ml LB培地に植菌し,培養した (37℃, 120rpm).18時間後,大腸菌を集菌し細胞破砕用バッファー
(50mM HEPES, 300mM NaCl, 1mM Tween
20, 1 tablet /10ml complete Mini EDTA) で細胞破砕した.その後,溶液にMagneHis Ni-Particles (Promega)
50µl を加え,緩く懸濁した.懸濁液は使用するまで-20℃で保存した.また、細胞抽出液による試験管内反応に用いた細胞抽出液は、同様に培養した大腸菌を超音波破砕したものを用いた。
試験管内反応
反応は精製したYhhYが付着しているビーズを洗浄用バッファー(50mM Tricine,20mM
KCl,5mM MgC2,0.2M MnCl,0.1mM Ca2+,0.1mM FeSO4,0.1mMZnSO4)で3回洗った後にPCRチューブで内行った.タンパクが付着したビーズ4µl,100mMの基質を4µlずつ,反応用バッファー(50mM Tricine,20mM
KCl,5mM MgC2,0.2M MnCl,0.1mM Ca2+,0.1mM FeSO4,0.1mMZnSO4,1mM MES,1mM 3AP)13.3µl,そこにDWを加えて計40µlになるように調製した.37℃で一晩振盪しながら反応させた.基質と生成物の定量にはLC/MSD 1100
Series (Agilent社)のSIMモードを使用した.測定条件はSaitoら(2006)の論文に従った.出力されたデータはMathematica7.0.1と積分用のスクリプトで内部標品に対する相対物質量を計算した.
形質転換
形質転換はケミカルコンピテントセルを用いて行った.大腸菌をLBプレートに画線培養し,コロニーを拾い2ml SOB培地 (tryptone 2g,
yeast extract 0.5g, Nacl 50mg, 0.4M KCl 625µl) に植菌し,30℃で培養した.一晩培養したら,培養液50µlを5ml SOB培地に植菌し,30℃で培養した.OD600が0.440になったら集菌し (3000rpm, 15分,4℃),培地を取り除いた.そのチューブに1.75mlの氷冷したTB液を加えた.さらに遠心し (3000rpm, 15分,4℃),0.4mlの氷冷したTB液で懸濁した.30µlのDMSOを加え,氷上に10分放置した.プラスミドを滅菌した2mlチューブの底に置き,氷上で3分冷やした.100µlコンピテントセルを加え,穏やかに混ぜ氷上で30分放置した.42℃で45秒ヒートショックを与え,氷上で30分放置した.0.9mlのSOC培地 (SOB培地10mlに対して20%グルコースを182µl加えた) を加え,37℃で1時間浸透せずに培養した.培養液100µlを30µg/ml chloramphenicolを含んだLB寒天培地で培養した.
菌体回収,メタボローム測定とデータ処理
メタボローム解析用の欠損株の回収は50ml M9培地が入った500mlフラスコを用いて37℃,120rpmで培養した.コバルトを添加する場合は15µM添加した,サンプリングの方法はOhashiらの方法に従った(Ohashi et al., 2008).培養液を0.45µmフィルター(Millipore)に通して集菌した.細胞を10ml Milli-Q水で2回洗った後,内部標品として2µM 2-(N-morpholino)ethanesulfonic acid, 2µM trimesate, 2µM methionine sulfone, 2µM 3-amino pyrrolidineが入った5mlメタノールにフィルターを浸して代謝を止めた.その後,5mlクロロホルムと1.6ml Milli-Q水を加え,よく混ぜた.混合液を遠心 (2300g,5分,4℃) し,4mlのメタノール層のみを5 kDaフィルター(Millipore)で高分子不純物を取り除いた.メタノールを蒸発させた後,測定まで-80℃で保存した.メタボローム測定の際に50µl Milli-Q水で乾燥した代謝物を溶かした.メタボローム測定はAgilent
CE capillary electrophoresis system (Agilent Technologies) を用いて行った.細かい機器設定はSogaらの方法に従った (Soga et al., 2006).すべての測定は3回ずつ別個に行った.出力されたデータはMaster Hands 2.11.0.2で積分した.Master Handsによってノイズだと判断されたピークを削除して,残ったピークについて解析を行った
結果と議論
YhhYタンパクによる3種のアミノ酸のアセチル化
大腸菌内で発現させた精製したYhhYタンパクを精製後にSDS-PAGEで確認したところ,目的の分子量のバンドが現れた (図 1) .また,目的以外のコンタミしたタンパクは肉眼で観察できなかった.次に,YhhYと20種のアミノ酸,アセチルCoAを反応させ副産物として生成されるCoAを定量した.その結果,特にメチオニン,ヒスチジンとフェニルアラニンがアセチル化された(図 2).さらにこの結果を追証するためにYhhYとメチオニン,ヒスチジン,フェニルアラニンとアセチルCoAを反応させた反応液をLC-MSで測定した.20種のアミノ酸をアセチル化して生成されうる物質のうち,YhhYを加えたサンプルと加えていないサンプル間に有意な差があった3物質の内部標品に対する相対物質量を図 3に示した.この実験からも3種のアミノ酸がYhhYによってアセチル化されたことが示された.これらの結果からYhhYは3種のアミノ酸をアセチル化する機能を持っていることが明らかになった.
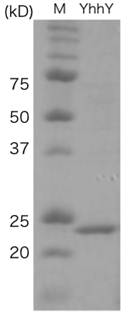
図 1組み換えタンパクの精製結果の確認
左はマーカー,右は精製した後のYhhYタンパク.15%
SDS-PAGEゲル,30mA,30分.
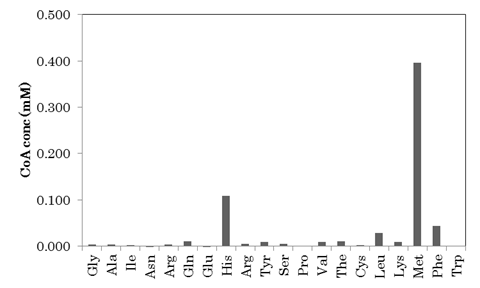
図 2 YhhY
の基質の探索
精製したYhhYと20種のアミノ酸,アセチルCoAを混合し37℃で一晩インキュベートしたときに,反応液に含まれるCoAをDTNBによって染色定量した
(n=1).
次に,メチオニン,ヒスチジンとフェニルアラニンのアセチル化反応が,コンタミした酵素ではなくYhhYによって引き起こされた反応であることを確認するために精製した酵素ではなく,YhhY過剰発現の細胞抽出液で試験管内反応を行った.もし図
1,図
2で示した反応がSDS-PAGEでバンドを確認できなかったコンタミした酵素によって引き起こされたならば,YhhYを過剰発現していない株の抽出液でもアセチル化反応が進むはずである.その結果,ヒスチジンとフェニルアラニンにおいてYhhY過剰発現株のみでアセチル化反応が起こったことから,これまで示してきた反応はYhhYによって引き起こされた可能性が高いことが確認できた(図
4).
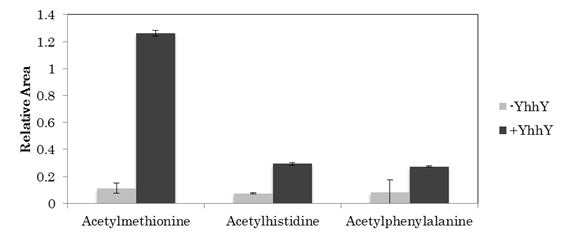
図 3 YhhYによって合成された3種の化合物
精製したYhhYとメチオニン,ヒスチジンとフェニルアラニンとアセチルCoAを混合し37℃で一晩インキュベートし生成物をLC-MSで測定したときの結果のうちYhhYを加えたサンプルと加えていないサンプル間に有意な差があった3物質の内部標品に対する相対物質量.灰色のバーは酵素を反応液に加えなかったもの (ネガティブコントロール)黒いバーはYhhYを加えたもの,縦軸は内部表品に対する相対面積.エラーバーは標準偏差(n=5).
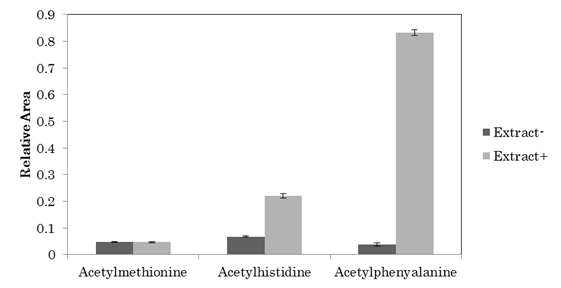
図 4 YhhY過剰発現株とコントロール株の細胞抽出液による試験管内
Cell extract of yhhY
overexpression strain was incubatd with methionine, histidine and phenylalanine (n=5).YhhY過剰発現株とコントロール株の細胞抽出液でメチオニン,ヒスチジンとフェニルアラニンをアセチル基受容体としてアセチル化反応を行った.エラーバーは標準偏差(n=5).
メチオニンを基質とした時のYhhYの基質親和性
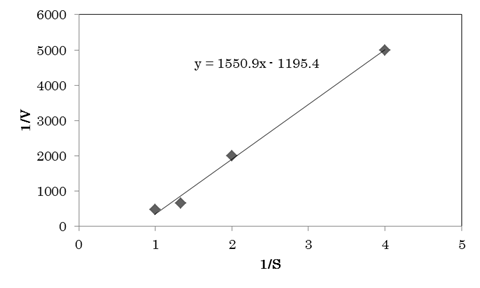
図 5 YhhYのメチオニンに対する反応速度
希釈系列を振ったメチオニン (2.5, 5.0, 7.5
1mM),と1mM アセチルCoAとYhhYを反応させたときの反応速度 (mM/min). 縦軸は反応速度V,横軸はメチオニン濃度.
次に,YhhYの基質との親和性を明らかにするために,酵素活性を定量するための条件検討を行った.今回はアセチルCoAの濃度は一定でメチオニンの濃度を変化させた.その結果,今回の基質濃度,酵素量,反応時間ではKmとVmaxが負の値になった(図 5).この問題を解決するためには酵素量や基質濃度を調整する必要がある.
チオール反応試薬によるYhhY酵素活性への影響
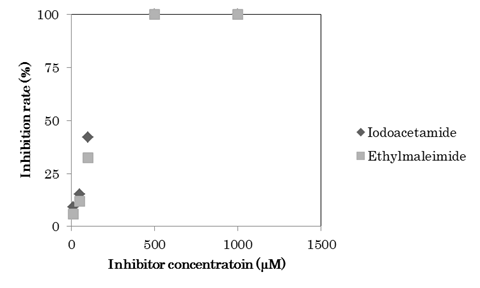
図 6 チオール反応試薬による酵素活性の阻害
ヨードアセトアミドとエチルマレイミドによるYhhYの酵素活性阻害率.横軸は阻害物質の濃度,縦軸は阻害率 (%).
新規酵素機能同定を行う上で,阻害物質の同定とその阻害率を明らかにすることは重要である.そこで,今回はチオール反応試薬としてよく知られているヨードアセトアミドとエチルマレイミドのYhhYに対する阻害活性を調べた.これらの試薬はアミノ酸のチオール基を不可逆的にアルキル化することで,ジスルフィド結合を阻害する.今回は,YhhYのメチオニンに対するアセチル化反応を,チオール反応試薬がどの程度阻害するのかを検証した.その結果,今回の実験条件では反応溶液に400µMのチオール反応試薬が存在するとYhhYの活性がほぼ無くなることが明らかになった(図
6).
最小培地におけるyhhY遺伝子欠損株とその親株のメタボローム解析
YhhYの生体内での働きを明らかにするため,yhhY欠損株のメタボローム解析を行った.また,今回のyhhY欠損株とその親株のメタボローム解析ではどちらからもYhhYがその合成に関わっていると考えられるアセチルメチオニン,アセチルヒスチジン,アセチルフェニルアラニンは検出されなかった.この結果から,今回の培養条件ではyhhY遺伝子が発現していなかったか,YhhYタンパクは低分子代謝に影響を及ぼしていいないという可能性が示唆された.その一方で,YhhYタンパクは遊離アミノ酸ではなく,ペプチドやタンパク中のアミノ酸をアセチル化している可能性も否定できない.もしYhhYタンパクがペプチドのN末端をアセチル化しているのであれば,そのペプチドは伸長することができなくなり,YhhYタンパクはタンパクの合成制御に関わっていることになる.また,原核生物のタンパクのアセチル化は高温耐性に関わっていることが明らかになっており,YhhYタンパクはストレス制御に関わる非常に重要な機能を持っている可能性がある(Ma et al., 2011).
yhhY遺伝子過剰発現株のメタボローム解析
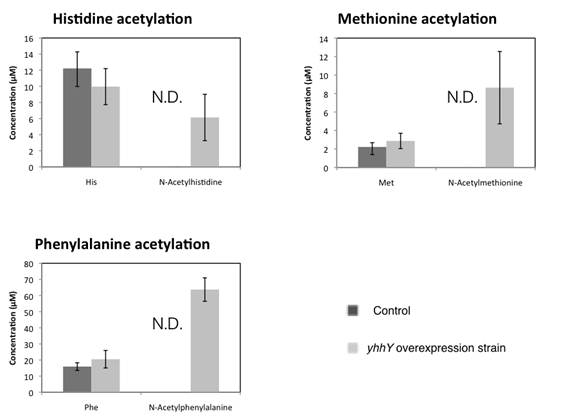
図
7 in vitroでアセチル化反応を確認できた3種のアミノ酸のyhhY遺伝子過剰発現株におけるin vivoでの蓄積量
YhhYがin vitroでアセチル化した3種のアミノ酸とその生産物であるアセチルアミノ酸の細胞内濃度.Aはヒスチジンのアセチル化反応.Bはメチオニンのアセチル化反応.Cはフェニルアラニンのアセチル化反応.黒のバーがコントロール株.灰色のバーがYhhYタンパク過剰発現株.エラーバーは標準偏差.反復実験は3回.N.D.は検出限界以下の物質.
試験管内でYhhYタンパクによるアセチル化が確認できた3種のアセチル化アミノ酸は,YhhY過剰発現株の細胞内でもその蓄積が確認できた(図 7).さらに,YhhYタンパクを過剰発現させていないコントロール株では全く蓄積されていないことから,アセチルヒスチジン,アセチルメチオニン,アセチルフェニルアラニンはYhhYタンパクによって生合成された可能性が高い.また,アセチルメチオニンとアセチルフェニルアラニンの基質であるメチオニンとフェニルアラニンは過剰発現株よりもコントロール株で細胞内濃度が高かった.これら2つのアミノ酸は過剰発現株内でアセチルメチオニンとアセチルフェニルアラニンの基質として利用されたために,コントロール株に比べて過剰発現株で細胞内濃度が減少したのだと考えられる.ヒスチジンについては,コントロール株の方が過剰発現株よりも細胞内濃度が低いが,アミノ酸は細胞内で幅広く用いられている物質であり,他の代謝経路の影響を受けて今回のような結果になった可能性がある.これらのことから,大腸菌のYhhYタンパクは生体内でも,3種のアセチル化アミノ酸の生合成の役割を担っていると考えられる.
謝辞
慶應義塾先端生命科学研究所Martin Robert特任講師には研究コンセプトの立案から,タンパクを扱う上での基本的な実験操作の指導まで大変お世話になりました.さらに,同研究所中東憲治准教授には遺伝学の立場から多くの有益なコメントを頂きました.最後に,同研究所冨田勝所長には素晴らしい研究環境を用意して頂きました.ここに厚く御礼申し上げます.
参考文献
Baba,T.
et al. (2006) Construction of Escherichia coli K-12 in-frame,single-gene
knockout mutants: the Keio collection. Mol.
Syst. Biol., 2, 2006.0008.
Carvalho,L.P.S.
de et al. (2010) Activity-based metabolomic profiling
of enzymatic function: identification of Rv1248c as a mycobacterial
2-hydroxy-3-oxoadipate synthase. Chem.
Biol., 17, 323-32.
Chen,L.
and Vitkup,D. (2007) Distribution of orphan metabolic
activities. Trends Biotechnol,25,
343-8.
Kanehisa,M.
and Goto,S. (2000) KEGG: kyoto
encyclopedia of genes and genomes. Nucleic
Acids Res., 28, 27-30.
Keseler,I.M.
et al. (2005) EcoCyc: a comprehensive database
resource for Escherichia coli. Nucleic
Acids Res., 33, D334-7.
Kitagawa,M.
et al. (2005) Complete set of ORF clones of Escherichia coli ASKA library (a
complete set of E. coli K-12 ORF archive): unique resources for biological
research. DNA Res., 12, 291-9.
Ma,Q.
and Wood,T.K. (2011) Protein acetylation in
prokaryotes increases stress resistance. Biochem. Biophys. Res. Commun.,
410, 846-51.
Ohashi,Y.
et al. (2008) Depiction of metabolome changes in histidine-starved Escherichia coli by CE-TOFMS. Mol Biosyst, 4,
135-47.
Saito,N.
et al. (2010) Unveiling cellular biochemical reactions via metabolomics-driven
approaches. Curr. Opin. Microbiol., 13, 358-62.
Saito,N.
et al. (2006) Metabolomics approach for enzyme discovery. J. Proteome Res.,5, 1979-87.
Saito,N.
et al. (2009) Metabolite profiling reveals YihU as a
novel hydroxybutyrate dehydrogenase for alternative
succinic semialdehyde metabolism in Escherichia coli.
J. Biol. Chem., 284, 16442-51.
Schnoes,A.M.
et al. (2009) Annotation error in public databases: misannotation
of molecular function in enzyme superfamilies. PLoS Comput. Biol.,
5, e1000605.
Soga,T.
et al. (2006) Differential metabolomics reveals ophthalmic acid as an oxidative
stress biomarker indicating hepatic glutathione consumption. J. Biol. Chem., 281, 16768-76.