| 3.結果 |
| 1.完全長cDNA配列とゲノム配列との比較 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 表2は、重複領域にマッピングされたcDNAをクラスタリングした際のcDNAのペア数・クラスター数の推移を示す。最終的に、O. sativa では674ク ラスター、M. musculus では107クラスターが導出された。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 表2:DNA領域に重複してマッピングされたcDNAクラスター | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
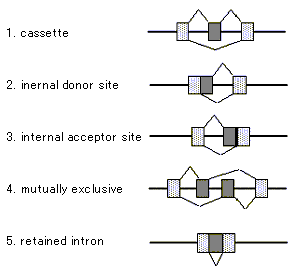
| また、各クラスターについて選択的スプライシングパターンを推定した。以下にスプライシングパターンの模式図[16]とともに、スプライスサイトの分類を示す。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
次に、興味深い特徴を示した結果をいくつか示す。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
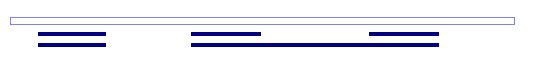
図5:Retained intron の例 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
選択的スプライシングパターンの一つに、イントロンがスプライスアウトされない例がある。下記の図に示されている例は、スプライスアウトされていないイントロンが、3'-末端付近に残っている。 cDNA生成過程にてunspliced なイントロンが残った結果であるかもしれないが、複数あるうちの1つのイントロンが残っているものが多かったため、これらのイントロンには何らかの機能が存在する可能性もあると考えられる。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||

図6:複数のエクソンが連続してスプライスアウトされる例 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
cDNA同士のアラインメントで確認されたギャップ領域に複数のエクソンが含まれていたことが、ゲノムにマッピングすることで確認された。下記の図にある例は、そのギャップ領域にエクソンが3つ含まれている。この領域のゲノム配列とcDNAの相同性が100%であることから、複数のエクソンが含まれている可能性が高いと言える。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||

図7:転写開始点がイントロン内にある例 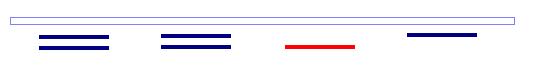
図8:転写終結点がイントロン内にある例 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
従来のcDNA同士の比較では導出が不可能であった 4.Mutually exclusive (異なるエキソンが選択されている選択的スプライシングパターン)のクラスターを導出することが可能となった。このパターンに分類された多くが、一方のcDNAのイントロン内にもう一方のcDNAの転写開始点や転写終結点が存在するというケースであり、非常に興味深い結果を得ることができた。 以上のような選択的スプラシングパターンに分類されたクラスターの他にも、一方のcDNAのイントロン内にもう一方のcDNA全体が組込まれているケースや、遺伝子領域がオーバーラッピングしているケースも含まれていた。また、5'末端側や3'末端側のエキソンの数が異なるcDNAクラスターも存在し、選択的プロモーターや選択的poly(A)の関与が示唆される。よって、完全長cDNA配列をゲノム配列にマッピングさせることによって、選択的スプライシング由来のcDNAの他にも 多様なcDNAクラスターを導出できることが示された。 2.マイクロアレイデータを用いた組織特異的cDNAの特定 これまでの研究においてマウスの完全長 21,000 cDNAs より、1,136本のcDNAからなる415クラスターを選択的スプライシング候補として導出した。このうち、マイクロアレイデータの発現パターンがある組織もしくはある発現段階でのみ異なっている4つのクラスターを同定した。以下にそのスプライシングパターンと発現量のグラフを示す。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||

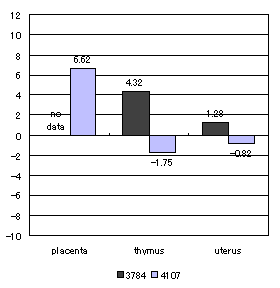
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
cluster ID:2204 にグルーピングされた2本のcDNAは、Prolaction-like-peptideと相同性が高かった。 Prolaction-like-peptideは prolaction (PRL)/growth hormone (GH) gene として知られており、pituitary gland, uterus や placenta にて発現することが報告されている[17]。図*より、seq ID: 4107は placenta で発現しているが thymus や uterusでは発現していないことが分かる。一方、Seq ID: 3784 は thymus や uterusにて高い発現量を示している。このことから、1つのエキソンが選択的にスプライスアウトされることによって、組織特異的に作用するタンパク質を作り出すことが考えられる。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
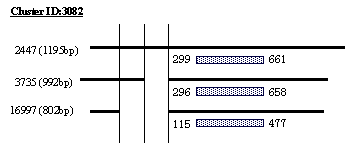
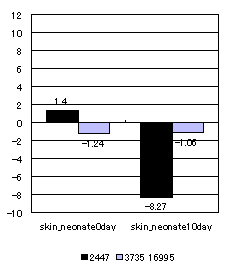
cluster ID:3082 にグルーピングされた3本のcDNAは、28S Ribosomal protein S17[18]と相同性が高かった。このクラスターでは、コード領域外の5'UTR領域において選択的スプライシングが行われており、タンパク質をコードするORF領域は3本ともに一致している。また、図*より、これらのcDNAはマウスの皮膚の成長過程においてそれぞれの発現量が段階的に制御されていることが分かる。5'UTRや3'UTRにおける選択的スプライシングは、遺伝子の発現量を制御する働きをする[3]ことから、この遺伝子は皮膚の発現段階に応じて選択的スプライシングによってRibosomal protein の発現量を変化させていることが示唆される。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
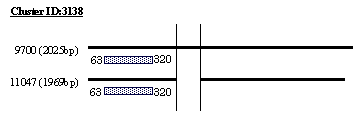
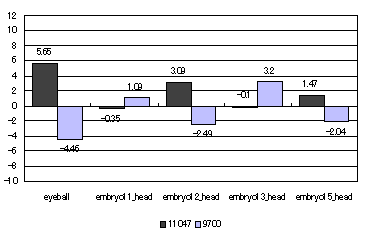
cluster ID: 3138 にグルーピングされた2本のcDNAは、TIA-1 cytotoxic granule-associated RNA-binding protein-like 1 と相同性が高かった。このタンパク質は、12.5日目の胎児のbrain や retinaで発現しており、また lung, kidney や thymus においても発現することが認められている[19]。cluster ID:3082 と同様、3'UTR領域において選択的スプライシングが行われることにより、胎児の脳の発育段階に応じて遺伝子の発現量が制御されている可能性がある。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
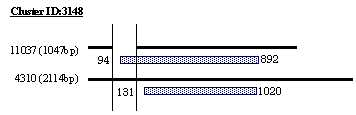
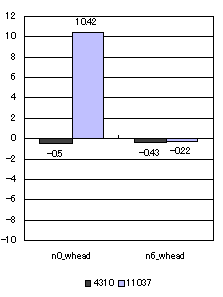
cluster ID: 3148 にグルーピングされた2本のcDNAは、bisphosphate 3'-nucleotidase [20]と相同性が高かった。このタンパク質は選択的スプライシングパターンをもつことは知られていない。今回見出されたスプライシングパターンによって、選択的にスプライスアウトされるエキソンの中にスタートコドンが存在し2種類のスタートコドンにて翻訳開始していることが予測される。 3.選択的スプライシングサイト周辺配列の解析 マウス及びヒトの既知選択的スプライシングによるmRNAを公共のデータベースであるGenBankより抽出し、前述の手法によりゲノム配列へのマッピングを行った。GenBankデータより抽出した既知選択的スプライシングによるmRNAの数と、ゲノム配列とのマッピングに成功したmRNAの数とを以下に示す。 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 表4:GenBankより抽出したmRNAの総数 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
M. musculus はマッピングに成功したmRNAの数が少なかったため、統計的に有意義な結論を出すのには不十分と判断されたことから、今回の解析ではH. sapiens のみを対象とした。選択的なスプライスサイト周辺配列と通常のスプライスサイト周辺配列の比較解析をするにあたり、各スプライスサイトの周辺8bpを抽出し、以下の表にまとめた。 また、スプライスサイト周辺配列は、以下に示されるコンセンサス配列に従うことが知られている[21]。
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
8bp around 5'-splice sites (Control Group)
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
8bp around 5'-splice sites (Alternatively Spliced)
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
8bp around 3'-splice sites (Control Group)
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
8bp around 3'-splice sites (Alternatively Spliced)
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 表5:スプライスサイト周辺配列の比較 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
上述の表を見る限り、選択的なスプライスサイトと通常のスプライスサイトは共にコンセンサス配列に従っているようである。また、それぞれのスプライスサイト周辺配列に統計的に有意義な差があるかを検証するため、カイ2乗検定を行った。結果、統計的に有意義な差はなく、選択的なスプサイスサイトと通常のスプライスサイトの周辺配列には違いがないという結論に達した。 |